df15_1 <- data.frame(
cho = c(5.68,3.79,6.02,4.85,4.60,6.05,4.90,7.08,3.85,4.65,4.59,4.29,7.97,
6.19,6.13,5.71,6.40,6.06,5.09,6.13,5.78,5.43,6.50,7.98,11.54,5.84,
3.84),
tg = c(1.90,1.64,3.56,1.07,2.32,0.64,8.50,3.00,2.11,0.63,1.97,1.97,1.93,
1.18,2.06,1.78,2.40,3.67,1.03,1.71,3.36,1.13,6.21,7.92,10.89,0.92,
1.20),
ri = c(4.53, 7.32,6.95,5.88,4.05,1.42,12.60,6.75,16.28,6.59,3.61,6.61,7.57,
1.42,10.35,8.53,4.53,12.79,2.53,5.28,2.96,4.31,3.47,3.37,1.20,8.61,
6.45),
hba = c(8.2,6.9,10.8,8.3,7.5,13.6,8.5,11.5,7.9,7.1,8.7,7.8,9.9,6.9,10.5,8.0,
10.3,7.1,8.9,9.9,8.0,11.3,12.3,9.8,10.5,6.4,9.6),
fpg = c(11.2,8.8,12.3,11.6,13.4,18.3,11.1,12.1,9.6,8.4,9.3,10.6,8.4,9.6,10.9,
10.1,14.8,9.1,10.8,10.2,13.6,14.9,16.0,13.2,20.0,13.3,10.4)
)
str(df15_1)
## 'data.frame': 27 obs. of 5 variables:
## $ cho: num 5.68 3.79 6.02 4.85 4.6 6.05 4.9 7.08 3.85 4.65 ...
## $ tg : num 1.9 1.64 3.56 1.07 2.32 0.64 8.5 3 2.11 0.63 ...
## $ ri : num 4.53 7.32 6.95 5.88 4.05 ...
## $ hba: num 8.2 6.9 10.8 8.3 7.5 13.6 8.5 11.5 7.9 7.1 ...
## $ fpg: num 11.2 8.8 12.3 11.6 13.4 18.3 11.1 12.1 9.6 8.4 ...
head(df15_1)
## cho tg ri hba fpg
## 1 5.68 1.90 4.53 8.2 11.2
## 2 3.79 1.64 7.32 6.9 8.8
## 3 6.02 3.56 6.95 10.8 12.3
## 4 4.85 1.07 5.88 8.3 11.6
## 5 4.60 2.32 4.05 7.5 13.4
## 6 6.05 0.64 1.42 13.6 18.311 多元线性回归
医学研究中许多疾病都有多种原因,而且预后也是由多种因素决定的。如糖尿病患者的血糖变化可能受胰岛素、糖化血红蛋白、血清总胆固醇、甘油三酯等多种生化指标的影响。即使对那些已知是由单一病原体导致的感染性疾病,也有许多因素影响是否发病,如遗传特征、感染途径及程度、自身免疫能力等。由于各因素间往往相互联系,多变量回归分析可以帮助我们分析变量间的数量依存关系,以及它们对结果变量的相对作用大小。多元线性回归(multiple linear regression),用于分析一个因变量与多个自变量之间的线性关系。
11.1 建立模型
使用一个医学中常见的数据进行演示:探索不同因素对空腹血糖的影响,数据录入如下:
数据一共5列,第1列是总胆固醇,第2列是甘油三酯,第3列是胰岛素,第4列是糖化血红蛋白,第5列是空腹血糖(因变量)。
在建立回归方程前,先简单探索下数据:
library(GGally)
## Warning: package 'GGally' was built under R version 4.3.3
ggpairs(df15_1) + theme_bw()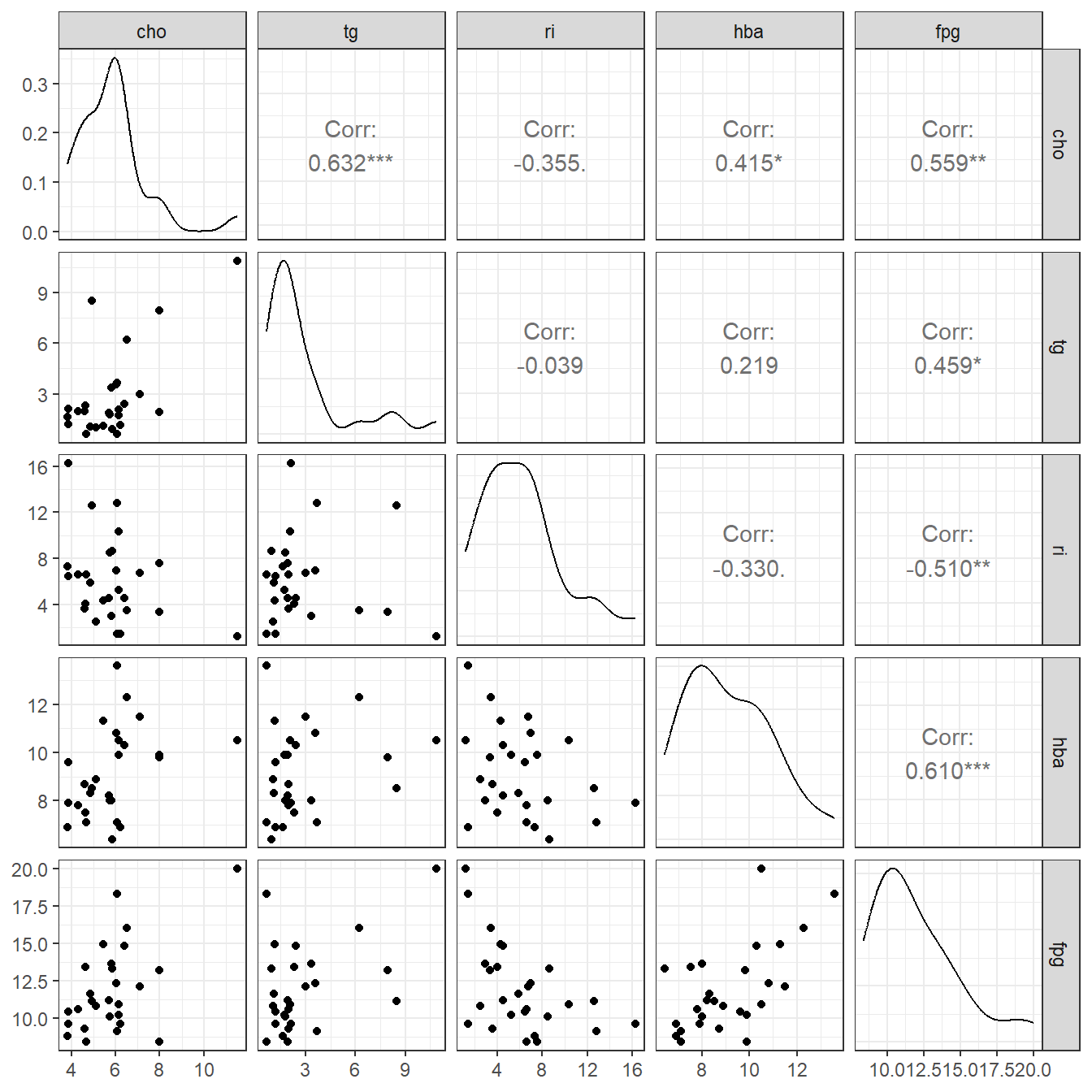
从这幅图来看,血糖和糖化血红蛋白相关性最大,和甘油三酯关系最小。
接下来建立回归方程:
f <- lm(fpg ~ cho + tg + ri + hba, data = df15_1)
summary(f)
##
## Call:
## lm(formula = fpg ~ cho + tg + ri + hba, data = df15_1)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.6268 -1.2004 -0.2276 1.5389 4.4467
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.9433 2.8286 2.101 0.0473 *
## cho 0.1424 0.3657 0.390 0.7006
## tg 0.3515 0.2042 1.721 0.0993 .
## ri -0.2706 0.1214 -2.229 0.0363 *
## hba 0.6382 0.2433 2.623 0.0155 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.01 on 22 degrees of freedom
## Multiple R-squared: 0.6008, Adjusted R-squared: 0.5282
## F-statistic: 8.278 on 4 and 22 DF, p-value: 0.0003121这个结果信息很丰富,给出了截距,各自变量的系数以及标准误、t值、P值,最下方给出了决定系数 R2,调整后的 R2,F值,总体方程的P值等。
11.2 模型评价
回归模型可以通过 R2、AIC、BIC、RMSE等评价,R2范围在0~1之间,越接近1说明结果越好。AIC、BIC、RMSE是越小越好。
library(performance)
r2(f)
## # R2 for Linear Regression
## R2: 0.601
## adj. R2: 0.528
AIC(f)
## [1] 120.78
BIC(f)
## [1] 128.5551
rmse(f)
## [1] 1.81395或者直接输出所有结果:
model_performance(f)
## # Indices of model performance
##
## AIC | AICc | BIC | R2 | R2 (adj.) | RMSE | Sigma
## ---------------------------------------------------------------
## 120.780 | 124.980 | 128.555 | 0.601 | 0.528 | 1.814 | 2.01011.3 回归诊断
判断数据是否满足多元线性回归的条件,也就是4个条件:
- 正态性
- 独立性
- 等方差性
- 线性
可以通过回归诊断图判断。
opar <- par(mfrow = c(2,2))
plot(f)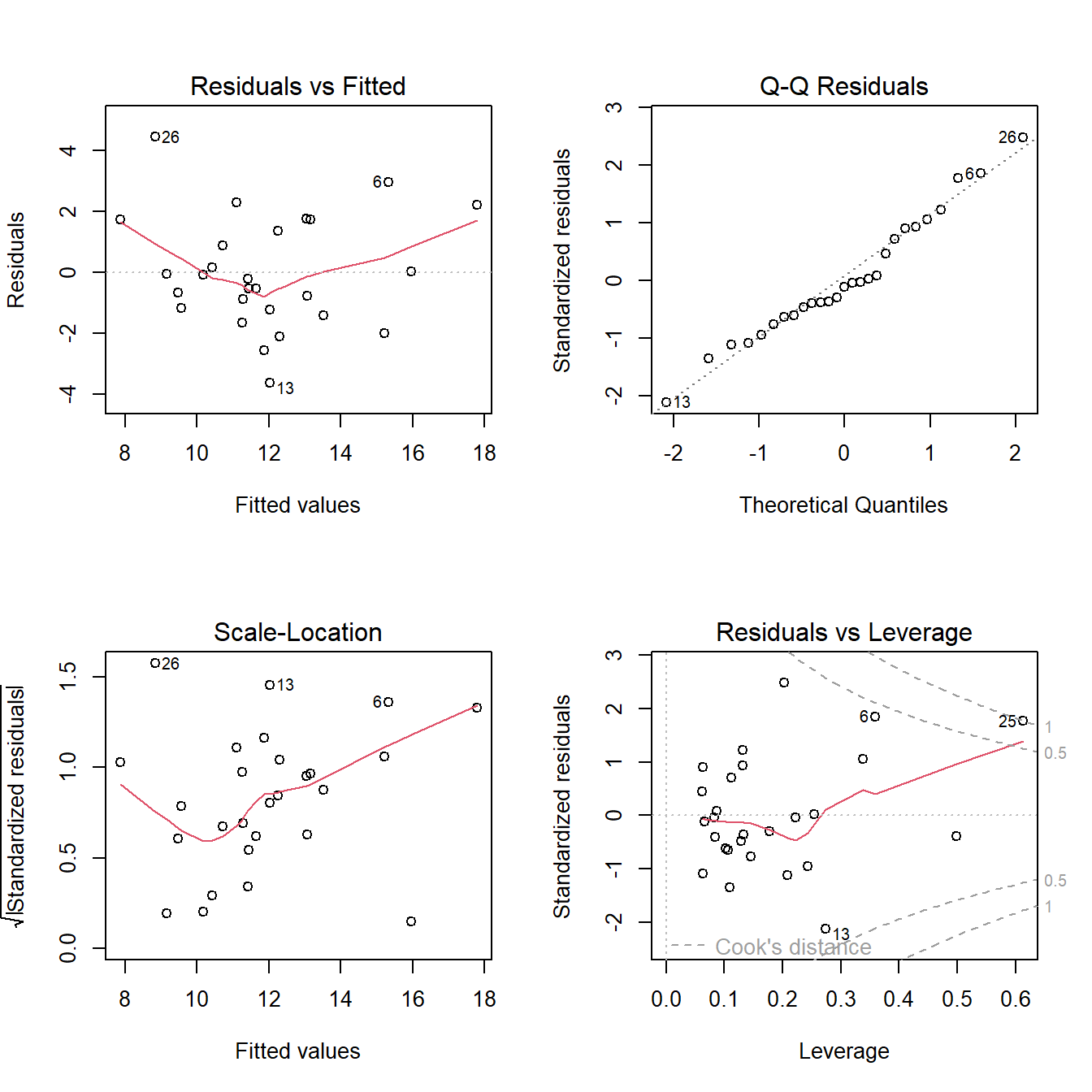
par(opar)第1幅图(左上)是残差拟合图,展示真实残差和拟合残差的关系,判读是否满足线性这个条件,如果满足,则应该为一条直线,但是本图明显是一条曲线,说明不是很满足线性这个条件,可能需要加二次项。
第2幅图(右上)是正态Q-Q图,判断是否满足正态性这个条件,通过这个图来看,基本满足。
第3幅图(左下)是位置尺度图,判读是否满足同方差性,如果满足,水平线两侧的点应该随机分布,从此图来看基本满足。
第4幅图(右下)是残差杠杆图,用于识别离群点等。
上面是比较原始的方法,下面介绍一个非常现代化的R包,用于实现以上图形:
library(performance)
check_model(f)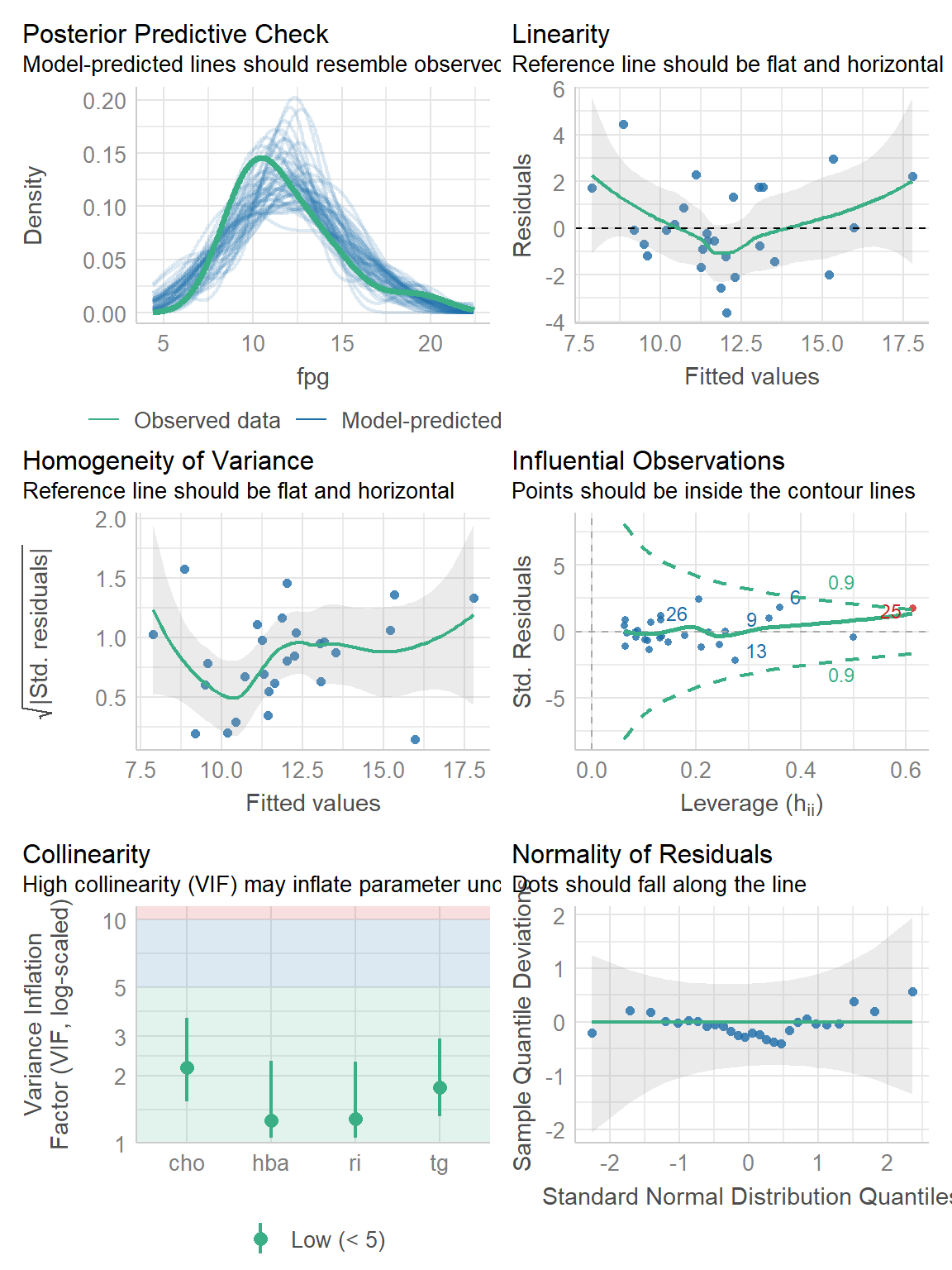
是不是更加好看了呢?
这几个图也可以单独画出来,使用以下代码即可:
diagnostic_plots <- plot(check_model(f, panel = FALSE))首先看第一个图。这个图是基于check_predictions()函数的,属于事后检验,是检查真实数据和模型数据的拟合情况的。下图中绿色粗线是真实的预测变量的分布情况,蓝色线条表示模拟的分布,理想的情况应该是完全重合的。从下图来看,其实是有些问题的,这说明我们用的模型可能不太合适。
diagnostic_plots[[1]]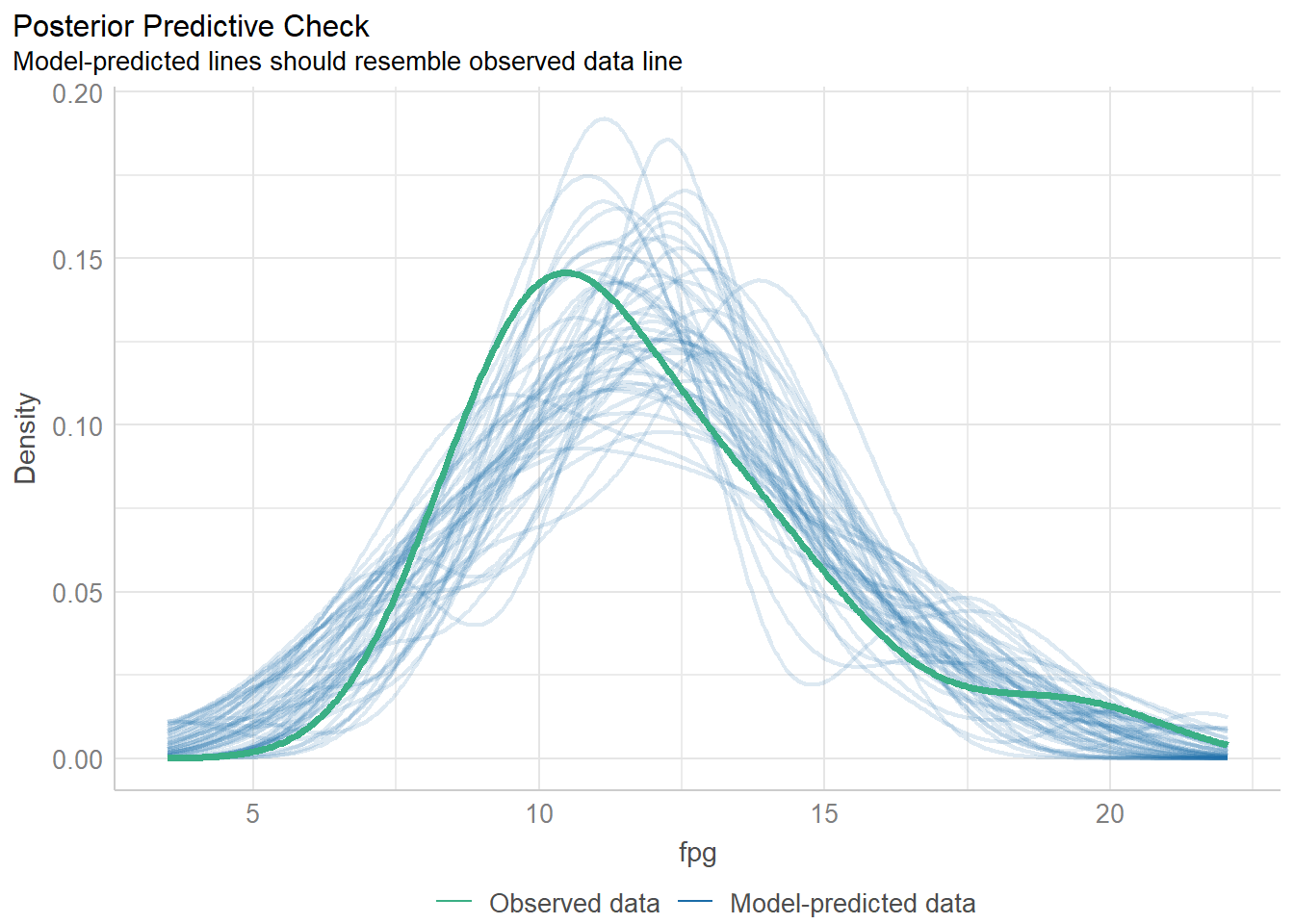
下面看第2张图。这张图是检查预测变量和结果变量是否符合线性关系的。合理的情况是残差完全随机地分布在参考线两侧。从这张图来看我们的数据其实不太完美。
diagnostic_plots[[2]]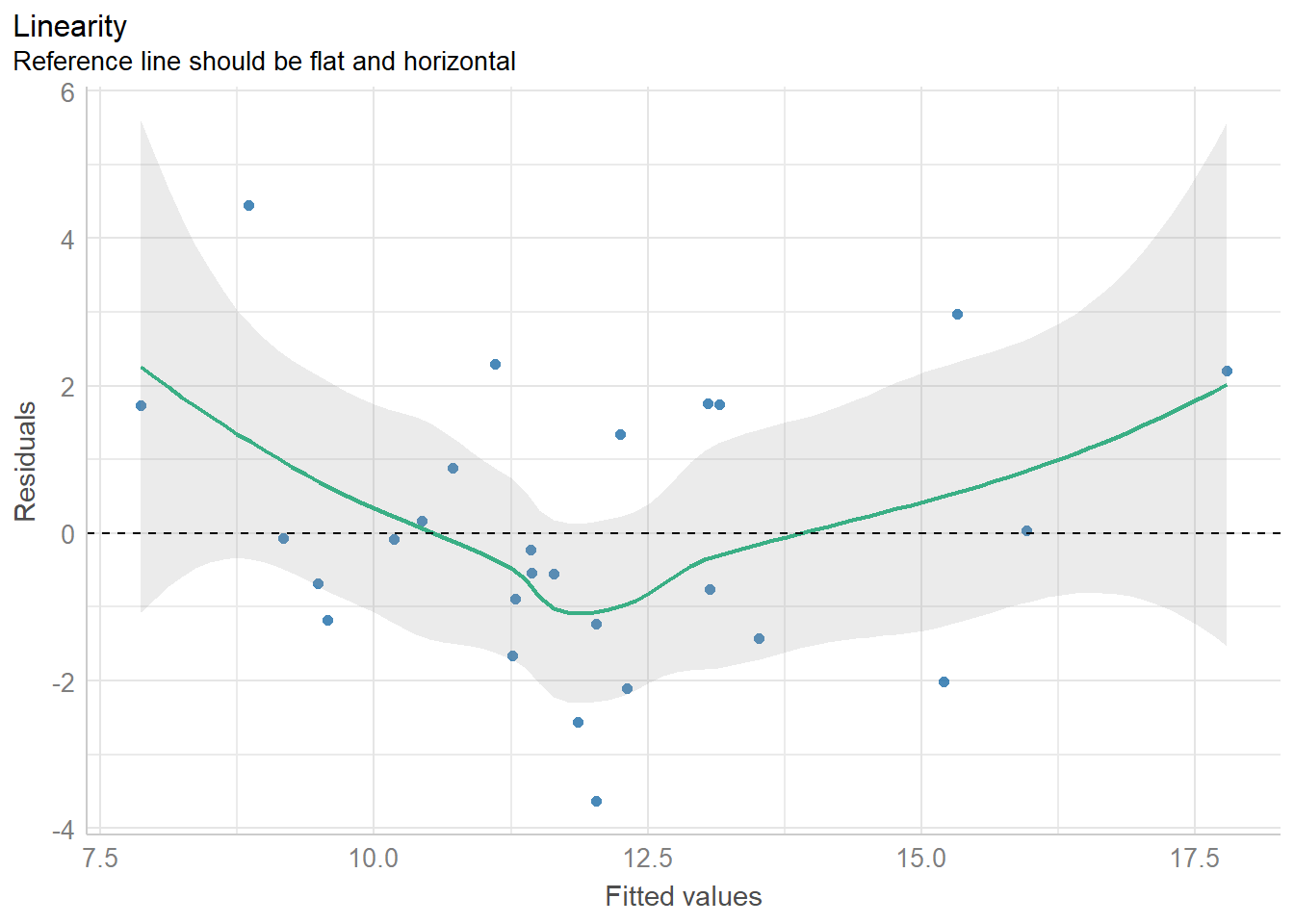
下面是第3幅图,是用来检查方差齐性的,同上面介绍过的位置尺度图。
diagnostic_plots[[3]]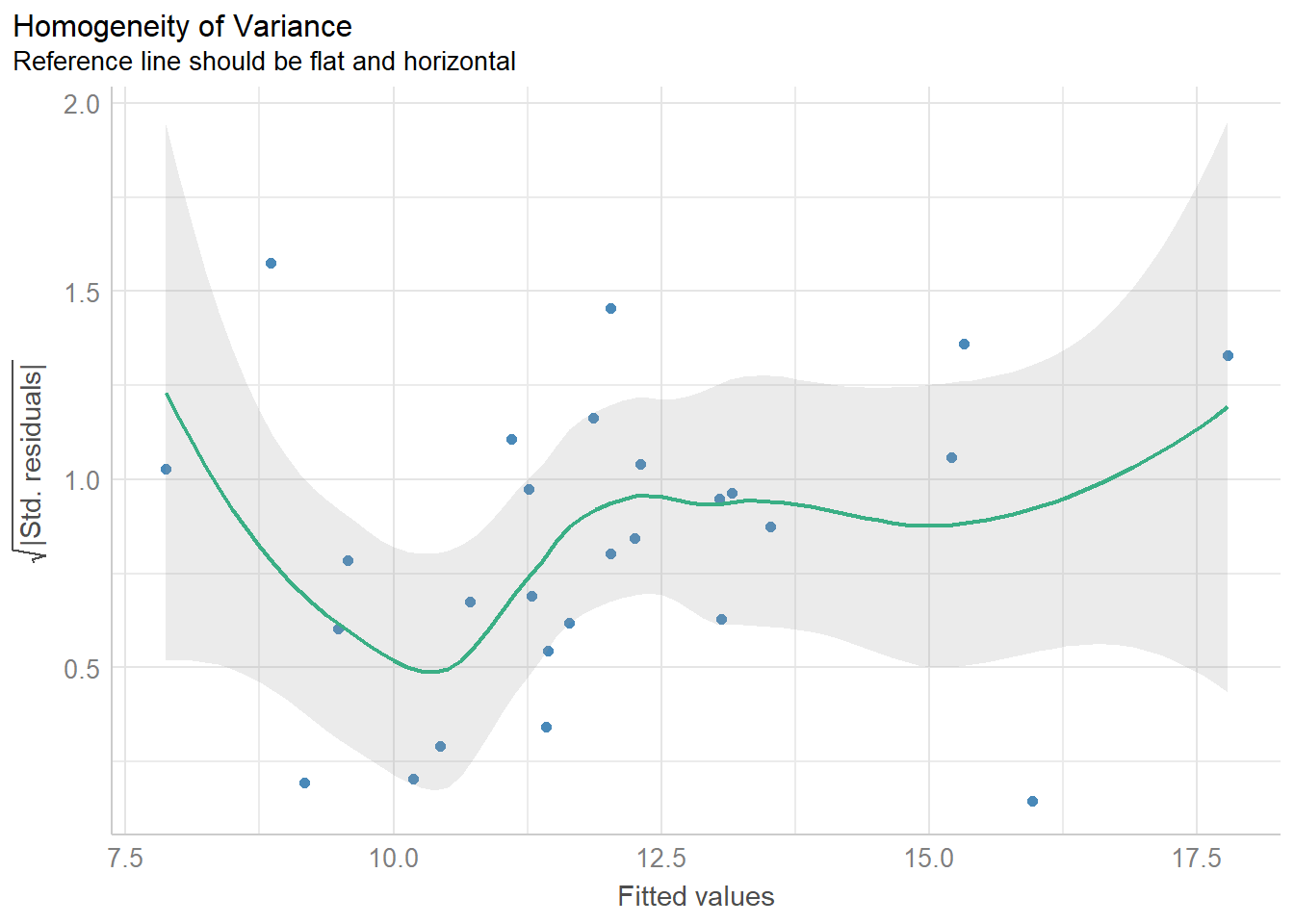
第4幅图是用来观察强影响点或者离群值、异常值的。使用的是库克距离(cook’s distance)来计算的,图中在虚线(库克距离)外的点可被认为是异常值。
diagnostic_plots[[4]]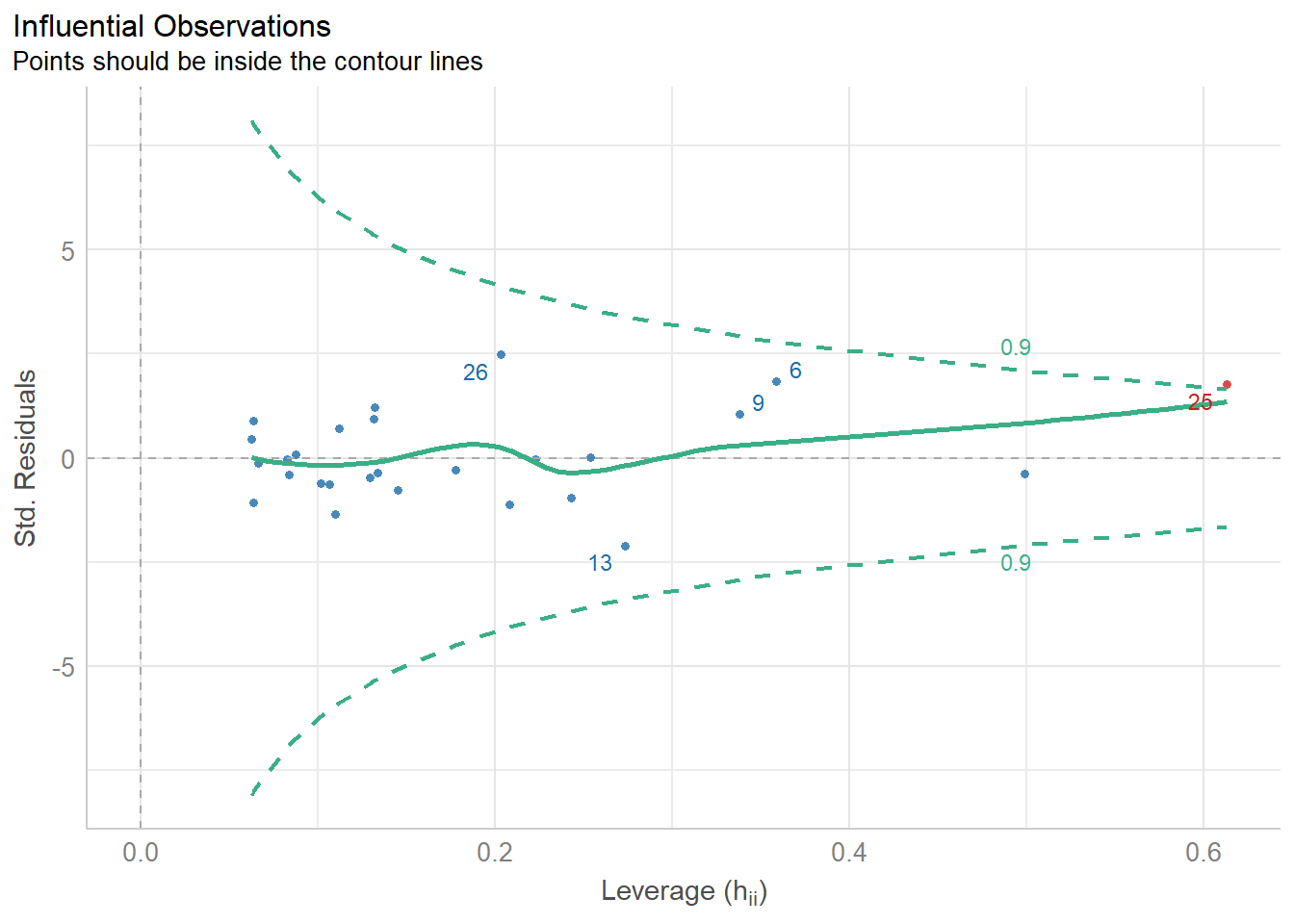
第5幅图是关于多重共线性的。是通过方差膨胀因子来评价的,下图中展示了4个变量的VIF,基本都在3以下,可认为不存在多重共线性:
diagnostic_plots[[5]]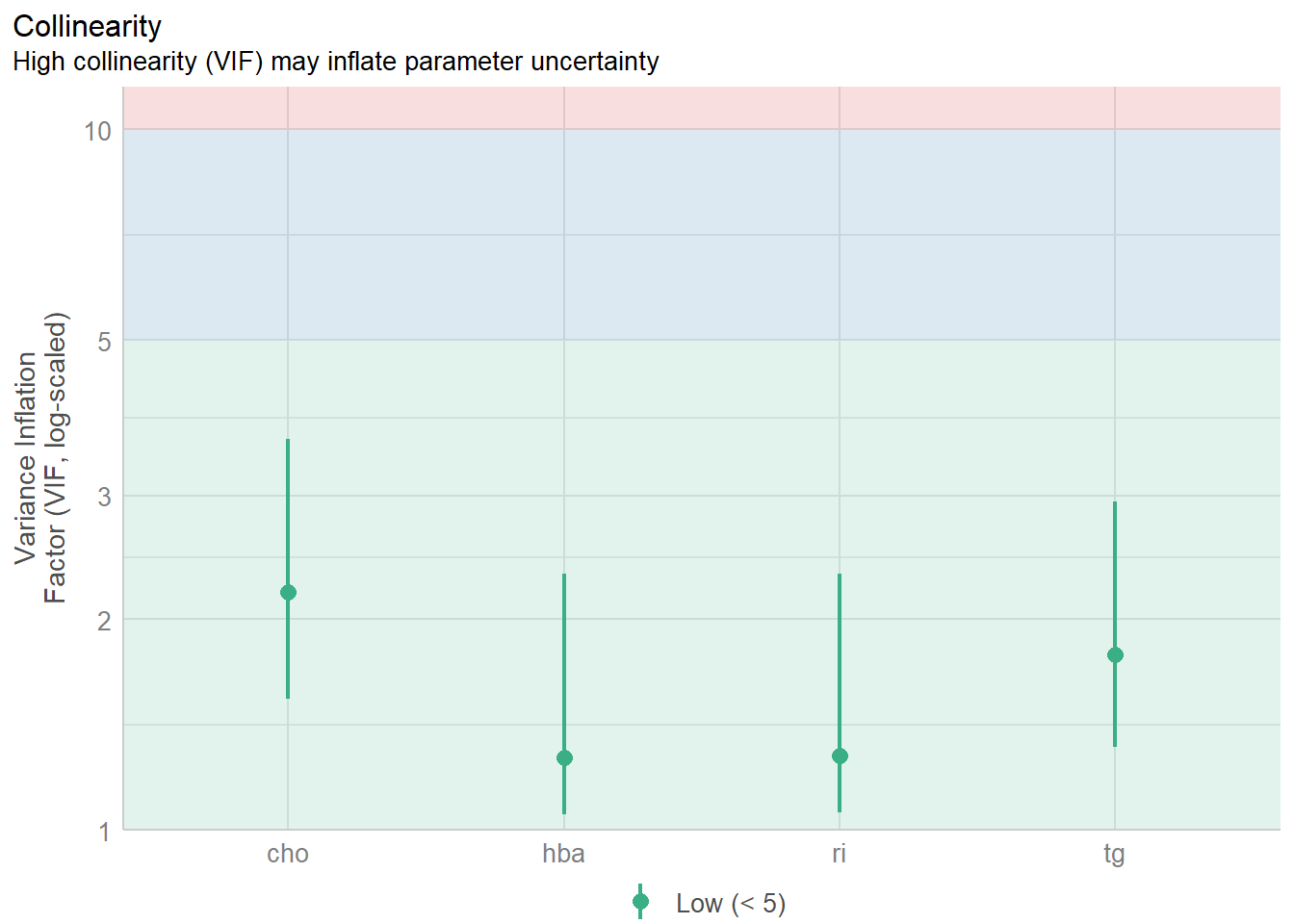
第6幅图是看正态性的。理想情况下数据点应该均匀的分布在横线上,最好是和横线重合,尤其是尾部,我们这个数据还算可以。
diagnostic_plots[[6]]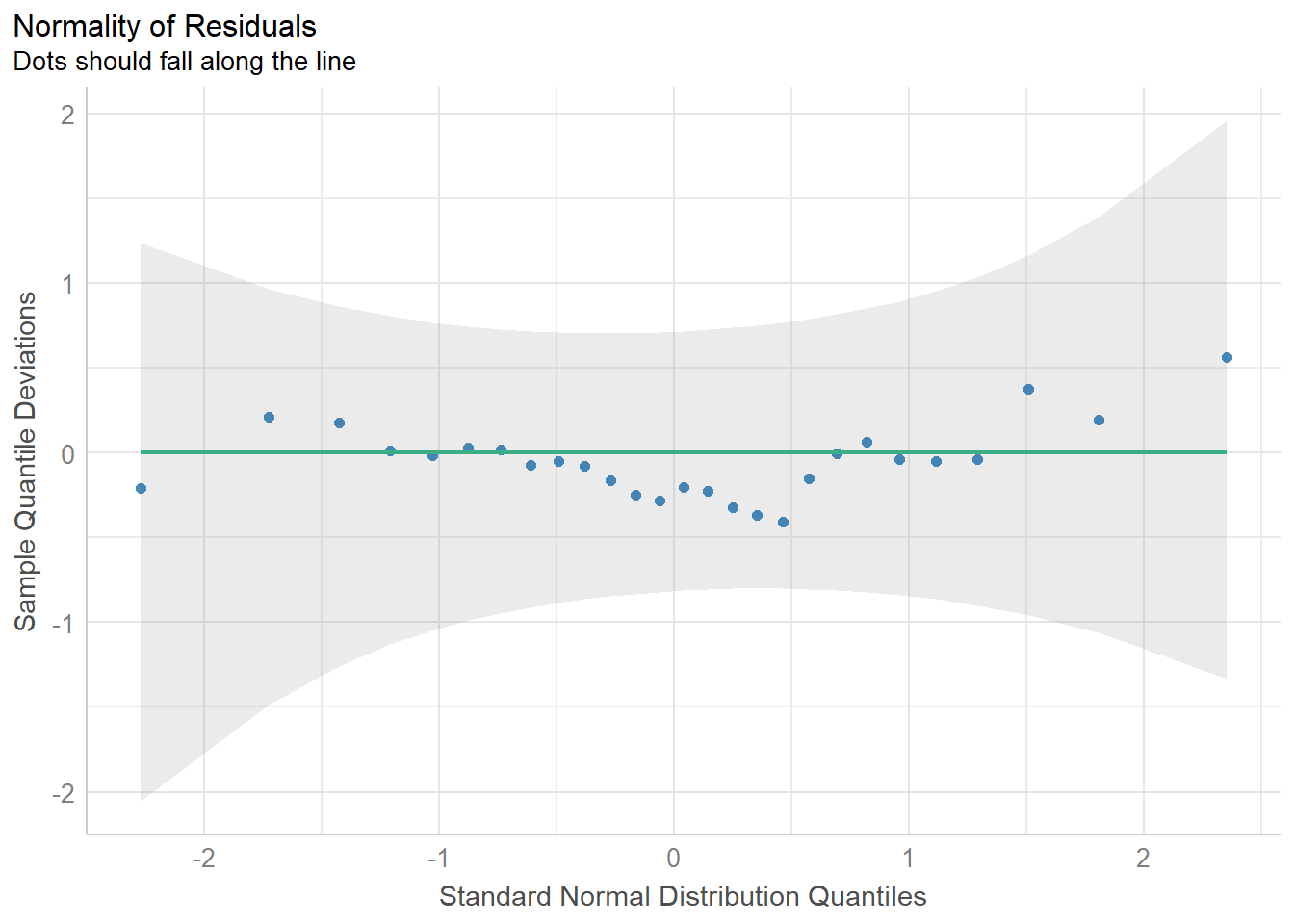
也可以通过统计方法判断,比如gvlma包可以实现对线性模型的综合判断:
library(gvlma)
gvmodel<-gvlma(f)
summary(gvmodel)
##
## Call:
## lm(formula = fpg ~ cho + tg + ri + hba, data = df15_1)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.6268 -1.2004 -0.2276 1.5389 4.4467
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.9433 2.8286 2.101 0.0473 *
## cho 0.1424 0.3657 0.390 0.7006
## tg 0.3515 0.2042 1.721 0.0993 .
## ri -0.2706 0.1214 -2.229 0.0363 *
## hba 0.6382 0.2433 2.623 0.0155 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.01 on 22 degrees of freedom
## Multiple R-squared: 0.6008, Adjusted R-squared: 0.5282
## F-statistic: 8.278 on 4 and 22 DF, p-value: 0.0003121
##
##
## ASSESSMENT OF THE LINEAR MODEL ASSUMPTIONS
## USING THE GLOBAL TEST ON 4 DEGREES-OF-FREEDOM:
## Level of Significance = 0.05
##
## Call:
## gvlma(x = f)
##
## Value p-value Decision
## Global Stat 9.68910 0.046003 Assumptions NOT satisfied!
## Skewness 0.65344 0.418886 Assumptions acceptable.
## Kurtosis 0.04015 0.841193 Assumptions acceptable.
## Link Function 7.68064 0.005582 Assumptions NOT satisfied!
## Heteroscedasticity 1.31487 0.251515 Assumptions acceptable.全局统计量:粗略估计结果变量和预测变量是否符合线性关系,结果是不符合 偏度和峰度:检验残差分布,结果是符合 连接函数:检测结果变量是连续型还是二分类,结果是不符合 异方差:检验方差齐性,结果是符合
以上是多个条件一起输出判断,也可以针对单独的条件进行判断。
首先看下正态性的判断。
library(car)
## Loading required package: carData
# 验证正态性
qqPlot(f,labels = row.names(df15_1), id.method = "identify",simulate = T,
main = "Q-Q plot") 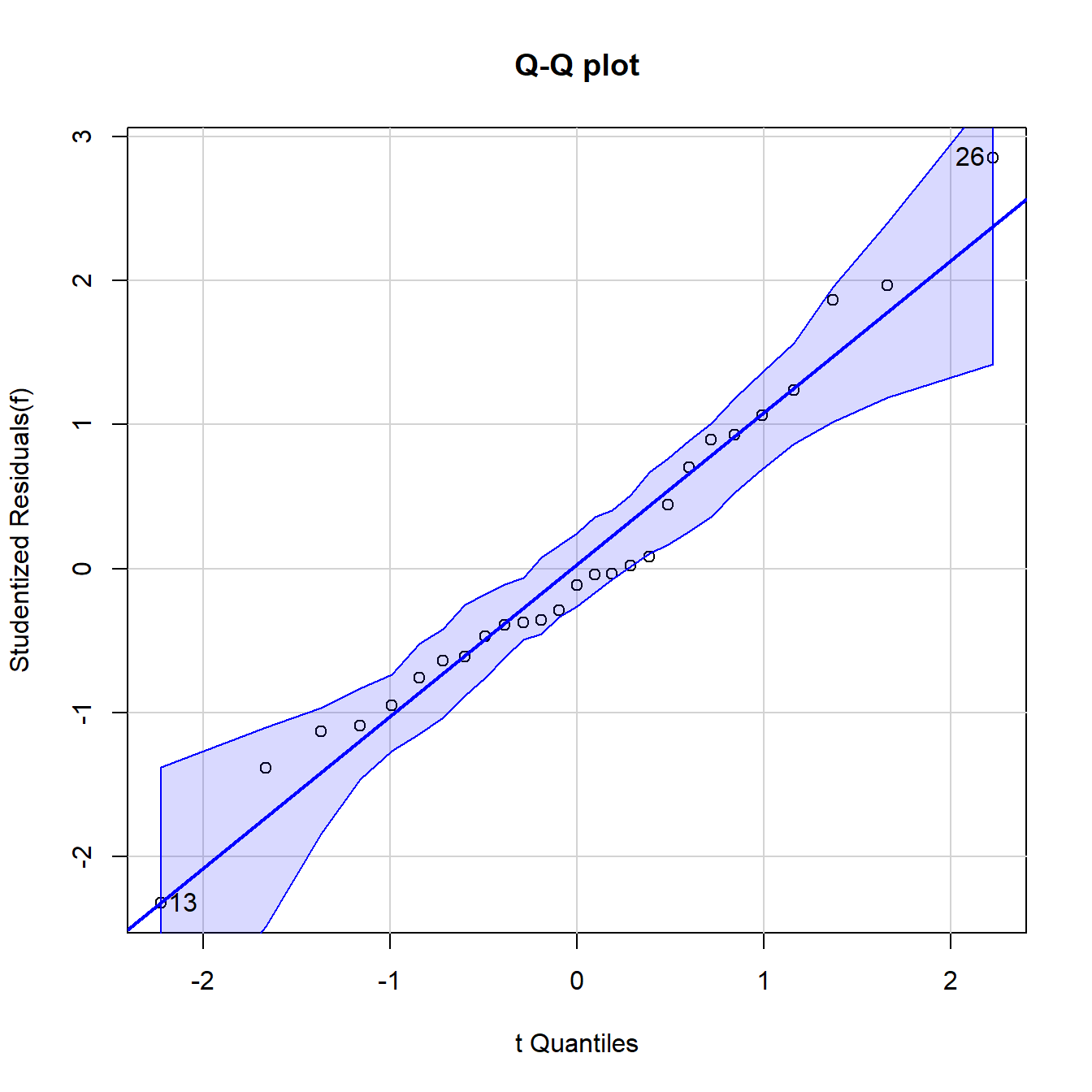
## [1] 13 26从图中可看出正态性基本满足。
当然也可以使用非常好用的performance包实现:
check_normality(f)
## OK: residuals appear as normally distributed (p = 0.671).检测离群值,基于cook距离:
check_outliers(f)
## 1 outlier detected: case 25.
## - Based on the following method and threshold: cook (0.9).
## - For variable: (Whole model).检测残差(或者因变量)独立性:
set.seed(123)
check_autocorrelation(f)
## OK: Residuals appear to be independent and not autocorrelated (p = 0.296).或者通过car包:
set.seed(123)
# 验证因变量独立性
durbinWatsonTest(f)
## lag Autocorrelation D-W Statistic p-value
## 1 0.1778885 1.634654 0.296
## Alternative hypothesis: rho != 0P值大于0.05,满足条件。
# 验证线性
crPlots(f)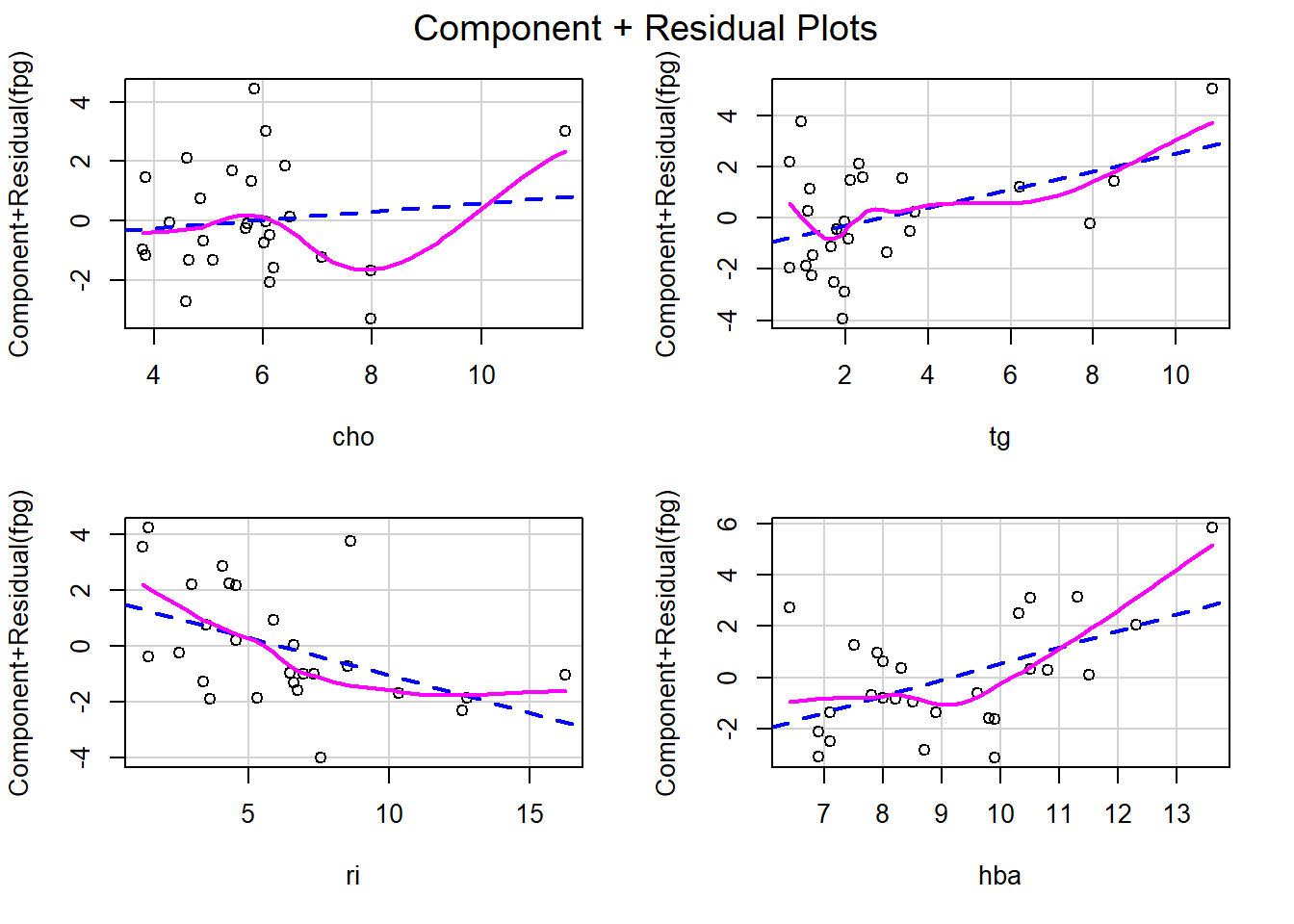
通过观察成分残差图,线性基本满足。
下面是检测方差齐性:
# 验证方差齐性
ncvTest(f)
## Non-constant Variance Score Test
## Variance formula: ~ fitted.values
## Chisquare = 0.0004274839, Df = 1, p = 0.9835P值大于0.05,方差齐性满足。
# performance检测方差齐性
check_heteroscedasticity(f)
## OK: Error variance appears to be homoscedastic (p = 0.984).结果一样的~
下面是多重共线性的检验,通过计算方差膨胀因子检验
vif(f)
## cho tg ri hba
## 2.185539 1.779862 1.278364 1.266730
vif(f)>4
## cho tg ri hba
## FALSE FALSE FALSE FALSE都小于4(标准有争议),基本不存在多重共线性。
或者通过performance实现:
check_collinearity(f)
## # Check for Multicollinearity
##
## Low Correlation
##
## Term VIF VIF 95% CI Increased SE Tolerance Tolerance 95% CI
## cho 2.19 [1.54, 3.62] 1.48 0.46 [0.28, 0.65]
## tg 1.78 [1.31, 2.95] 1.33 0.56 [0.34, 0.76]
## ri 1.28 [1.06, 2.32] 1.13 0.78 [0.43, 0.94]
## hba 1.27 [1.05, 2.32] 1.13 0.79 [0.43, 0.95]11.4 参考资料
- R语言实战
- 孙振球版医学统计学第四版
- performance包官方文档