df <- read.csv("datasets/例20-1.csv")
psych::headTail(df)
## id x1 x2 x3 y
## 1 1 23 8 0 1
## 2 2 -1 9 -2 1
## 3 3 -10 5 0 1
## 4 4 -7 -2 1 1
## ... ... ... ... ... ...
## 19 19 -9 -20 3 2
## 20 20 -7 -2 3 2
## 21 21 -9 6 0 2
## 22 22 12 0 0 227 判别分析
判别分析(discriminant-analysis)是根据判别对象若干个指标的观测结果判定其属于哪一类的统计方法。经典的判别分析方法有Fisher判别和贝叶斯判别分析。当分类很确定时,判别分析可以有效替代logistic回归,但是如果自变量和因变量关系很复杂时,判别分析表现不如logistic回归。
27.1 Fisher判别分析
Fisher判别又称为典型判别(canonical discriminant)分析,适用于两类和多分类判别。
Fisher判别使用贝叶斯定理确定每个观测属于某个类别的概率。如果你有两个类别,比如良性和恶性,判别分析会分别计算属于两个类别的概率,然后选择概率大的类别作为正确的类别。
线性判别分析假设每个类中的观测服从多元正态分布,并且不同类别之间的协方差相等。二次判别假设观测服从正态分布,每种类别都有自己的协方差。
使用孙振球版《医学统计学》第4版例20-1的数据。电子版及配套数据已上传到QQ群,需要的加群下载即可。
收集了22例肝硬化患者的3个指标,其中早期患者(用1表示)12例,晚期患者(用2表示),试做判别分析。
这个数据集中id是编号,x1,x2,x3是自变量,y是因变量。
线性判别分析可以通过MASS包中的lda函数实现:
library(MASS)
fit <- lda(y ~ x1+x2+x3, data = df)
fit
## Call:
## lda(y ~ x1 + x2 + x3, data = df)
##
## Prior probabilities of groups:
## 1 2
## 0.5454545 0.4545455
##
## Group means:
## x1 x2 x3
## 1 -3 4 -1
## 2 4 -5 1
##
## Coefficients of linear discriminants:
## LD1
## x1 0.0395150
## x2 -0.1265698
## x3 0.1792631Prior probabilities of groups是先验概率,类别1的概率是0.5454545,类别2是0.4545455。
然后给出了每个组在不同类别中的均值。
最下面给出了线性判别系数,如果你的结果变量是3个类别,会给出两组判别系数,这里我的结果变量只有2分类,所以结果只有1组。
结果可以画出来:
plot(fit,type="both")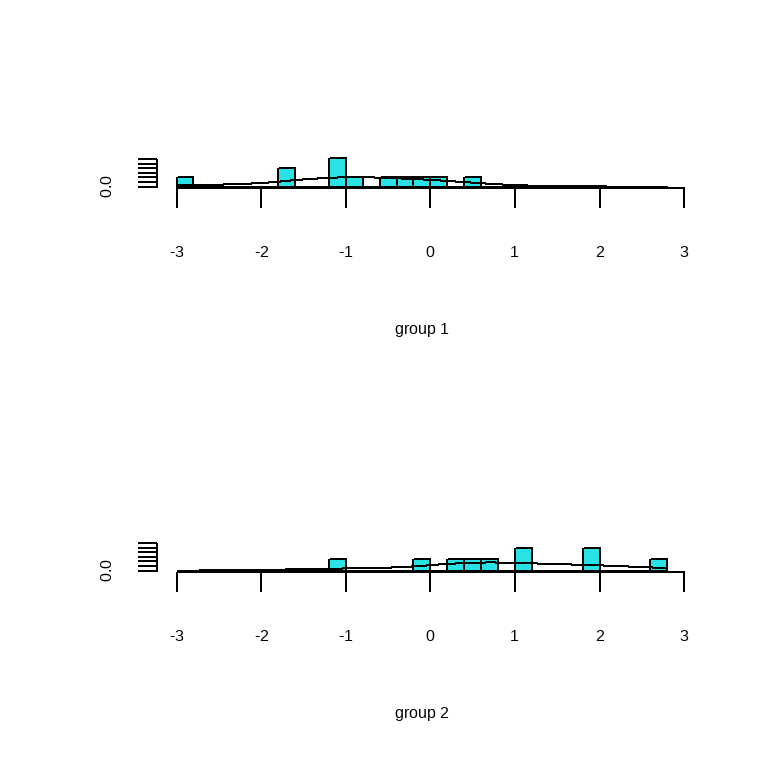
上图是判别分析结果的直方图和密度图,可以看出组间有重合,说明有些分组分错了。
下面用predict提取判别分析的分类结果。
predict用于判别分析可以得到3种类型的结果,class是类别,posterior是概率,x是线性判别评分。
pred <- predict(fit)$class
table(df$y, pred)
## pred
## 1 2
## 1 11 1
## 2 2 8可以看到有3个分类分错了,结果还是可以的。
可以查看每个患者的后验概率:
# 查看概率
predict(fit)$posterior
## 1 2
## 1 0.62566758 0.374332416
## 2 0.95508370 0.044916302
## 3 0.89600449 0.103995511
## 4 0.51330556 0.486694443
## 5 0.95464457 0.045355435
## 6 0.88314148 0.116858515
## 7 0.77454260 0.225457398
## 8 0.99508599 0.004914013
## 9 0.89391137 0.106088634
## 10 0.84899794 0.151002059
## 11 0.31960372 0.680396284
## 12 0.64144092 0.358559076
## 13 0.14903037 0.850969632
## 14 0.57026493 0.429735074
## 15 0.13106732 0.868932682
## 16 0.26925350 0.730746503
## 17 0.03911397 0.960886034
## 18 0.04332382 0.956676176
## 19 0.01115243 0.988847571
## 20 0.35826933 0.641730670
## 21 0.90954200 0.090457999
## 22 0.37480490 0.625195100上面的图我们也可以用ggplot2画出来。
df.plot <- data.frame(LD1 = predict(fit)$x[,1],
y = factor(df$y,labels = c("早期患者","晚期患者"))
)
library(ggplot2)
ggplot(df.plot, aes(x=LD1, fill=y))+
geom_histogram()+
facet_wrap(~ y, ncol = 1)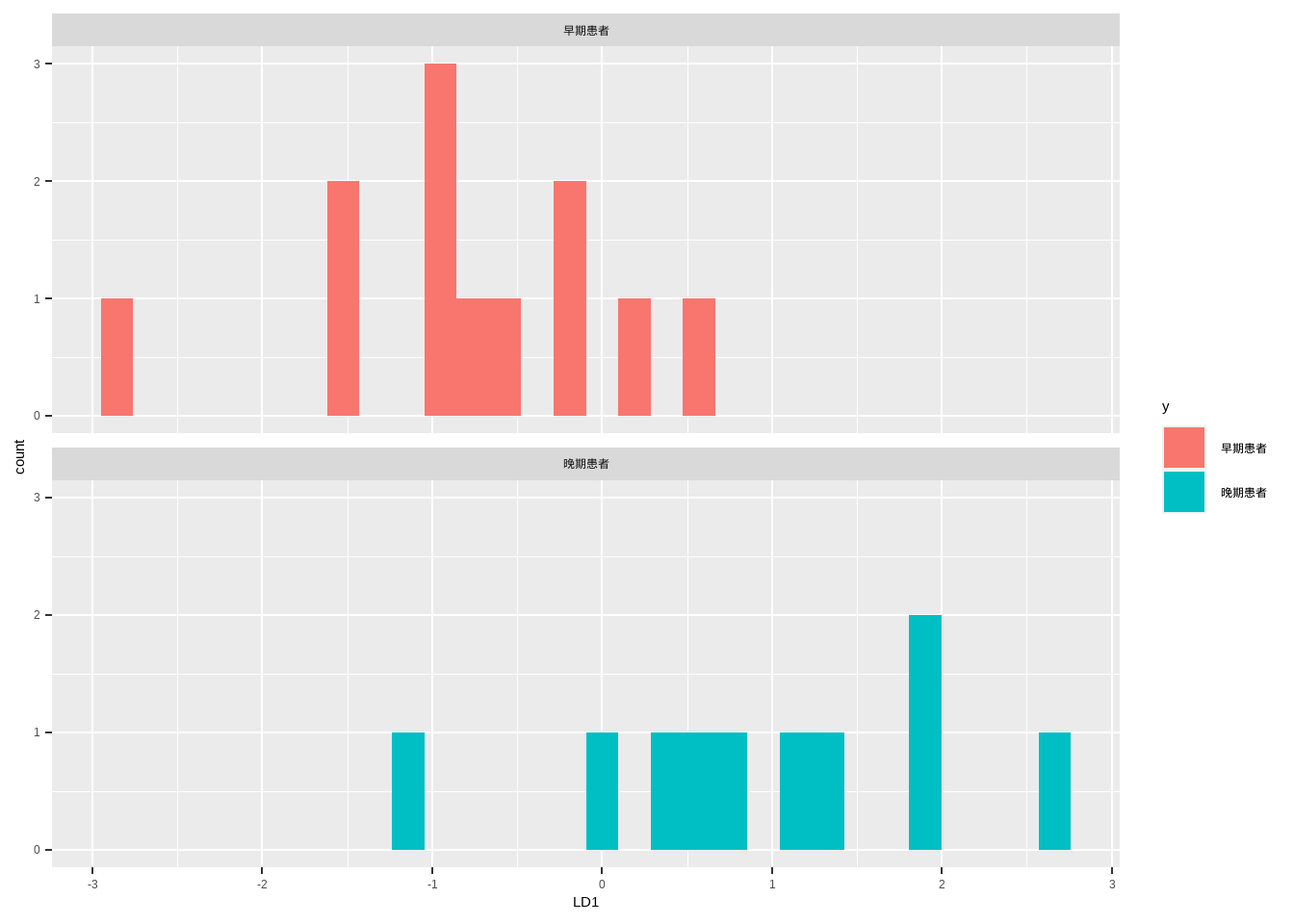
如果你想用这个模型预测新的数据,只需要predict(fit, newdata = xxx)即可。比如我们新建一个数据:
tmp <- data.frame(x1 = c(-9,-7,-9),
x2 = c(-18,-2,6),
x3 = c(3,3,1)
)
predict(fit, newdata = tmp)
## $class
## [1] 2 2 1
## Levels: 1 2
##
## $posterior
## 1 2
## 1 0.01736557 0.9826344
## 2 0.35826933 0.6417307
## 3 0.87974275 0.1202573
##
## $x
## LD1
## 1 2.4580167
## 2 0.5119296
## 3 -0.9381851这样就得到新的结果。
我们再用一个iris鸢尾花数据集演示下线性判别分析的结果可视化,这个结果变量是3分类的。
str(iris)
## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...拟合模型:
library(MASS)
fit <- lda(Species ~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width, data = iris)
fit
## Call:
## lda(Species ~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width,
## data = iris)
##
## Prior probabilities of groups:
## setosa versicolor virginica
## 0.3333333 0.3333333 0.3333333
##
## Group means:
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## setosa 5.006 3.428 1.462 0.246
## versicolor 5.936 2.770 4.260 1.326
## virginica 6.588 2.974 5.552 2.026
##
## Coefficients of linear discriminants:
## LD1 LD2
## Sepal.Length 0.8293776 -0.02410215
## Sepal.Width 1.5344731 -2.16452123
## Petal.Length -2.2012117 0.93192121
## Petal.Width -2.8104603 -2.83918785
##
## Proportion of trace:
## LD1 LD2
## 0.9912 0.0088可视化结果:
iris$LD1 <- predict(fit)$x[,1]
iris$LD2 <- predict(fit)$x[,2]
library(ggplot2)
ggplot(iris, aes(LD1,LD2))+
geom_point(aes(color=Species),size=3)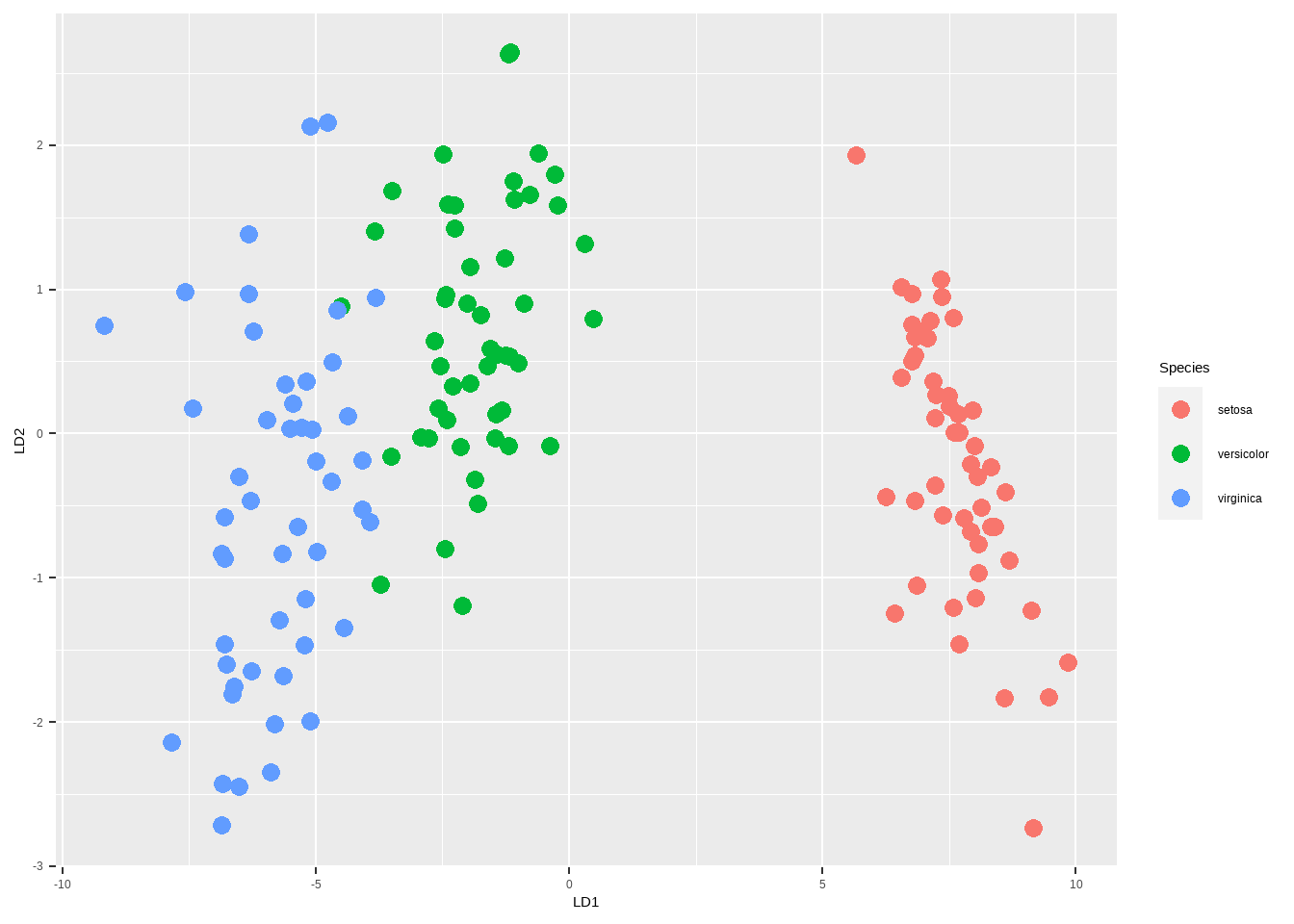
ggplot(iris, aes(x=LD1, fill=Species))+
geom_histogram()+
facet_wrap(~ Species, ncol = 1)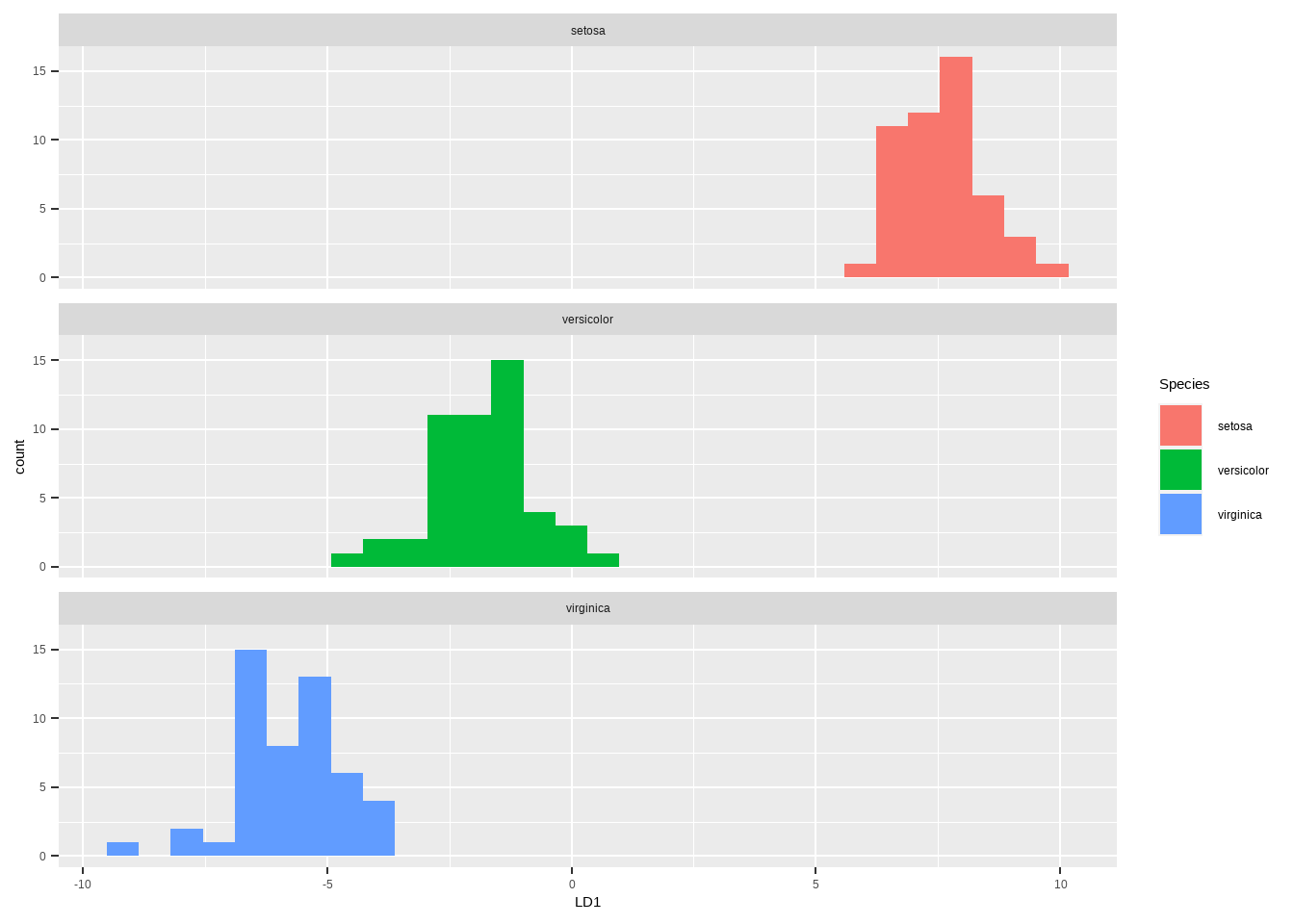
二次判别分析和线性判别分析用法一样。
fit <- qda(y ~ x1+x2+x3, data = df)
fit
## Call:
## qda(y ~ x1 + x2 + x3, data = df)
##
## Prior probabilities of groups:
## 1 2
## 0.5454545 0.4545455
##
## Group means:
## x1 x2 x3
## 1 -3 4 -1
## 2 4 -5 1结果不含判别系数,查看分类结果:
pred <- predict(fit)$class
table(df$y, pred)
## pred
## 1 2
## 1 10 2
## 2 1 9也是3个分错了。
27.2 Bayes判别分析
贝叶斯判别也是根据概率大小进行判别,要求各类近似服从多元正态分布。当各类的协方差相等时,可得到线性贝叶斯判别函数,当各类的协方差不相等时,可得到二次贝叶斯判别函数。
欲用4个标化后的影像学指标鉴别脑囊肿(1)、胶质瘤(2)、转移瘤(3),收集了17个病例,试建立判别贝叶斯函数。
df <- read.csv("datasets/例20-4.csv")
df$y <- factor(df$y)
psych::headTail(df)
## x1 x2 x3 x4 y
## 1 6 -11.5 19 90 1
## 2 -11 -18.5 25 -36 3
## 3 90.2 -17 17 3 2
## 4 -4 -15 13 54 1
## ... ... ... ... ... <NA>
## 14 10 -18 14 50 1
## 15 -8 -14 16 56 1
## 16 0.6 -13 26 21 3
## 17 -40 -20 22 -50 3使用klaR包实现贝叶斯判别分析:
library(klaR)
fit <- NaiveBayes(y ~ ., data = df)
fit
## $apriori
## grouping
## 1 2 3
## 0.4117647 0.2352941 0.3529412
##
## $tables
## $tables$x1
## [,1] [,2]
## 1 -14.42857 38.26163
## 2 0.80000 78.10779
## 3 -6.65000 19.78017
##
## $tables$x2
## [,1] [,2]
## 1 -17.34286 4.103599
## 2 -17.42500 3.085855
## 3 -17.33333 4.143268
##
## $tables$x3
## [,1] [,2]
## 1 12.71429 4.990467
## 2 17.50000 2.081666
## 3 20.16667 6.493587
##
## $tables$x4
## [,1] [,2]
## 1 31.14286 44.03948
## 2 0.00000 30.75711
## 3 -15.00000 35.83295
##
##
## $levels
## [1] "1" "2" "3"
##
## $call
## NaiveBayes.default(x = X, grouping = Y)
##
## $x
## x1 x2 x3 x4
## 1 6.0 -11.5 19 90
## 2 -11.0 -18.5 25 -36
## 3 90.2 -17.0 17 3
## 4 -4.0 -15.0 13 54
## 5 0.0 -14.0 20 35
## 6 0.5 -11.5 19 37
## 7 -10.0 -19.0 21 -42
## 8 0.0 -23.0 5 -35
## 9 20.0 -22.0 8 -20
## 10 -100.0 -21.4 7 -15
## 11 -100.0 -21.5 15 -40
## 12 13.0 -17.2 18 2
## 13 -5.0 -18.5 15 18
## 14 10.0 -18.0 14 50
## 15 -8.0 -14.0 16 56
## 16 0.6 -13.0 26 21
## 17 -40.0 -20.0 22 -50
##
## $usekernel
## [1] FALSE
##
## $varnames
## [1] "x1" "x2" "x3" "x4"
##
## attr(,"class")
## [1] "NaiveBayes"获取预测结果,并查看混淆矩阵:
pred <- predict(fit)$class
## Warning in FUN(X[[i]], ...): Numerical 0 probability for all classes with
## observation 10
table(pred, df$y)
##
## pred 1 2 3
## 1 7 0 1
## 2 0 3 0
## 3 0 1 5只有两个分错了。
如果要预测新的数据,只需要predict(fit, newdata = xxx)即可。