library(survival)
rm(list = ls())
dim(lung)
## [1] 228 10
str(lung)
## 'data.frame': 228 obs. of 10 variables:
## $ inst : num 3 3 3 5 1 12 7 11 1 7 ...
## $ time : num 306 455 1010 210 883 ...
## $ status : num 2 2 1 2 2 1 2 2 2 2 ...
## $ age : num 74 68 56 57 60 74 68 71 53 61 ...
## $ sex : num 1 1 1 1 1 1 2 2 1 1 ...
## $ ph.ecog : num 1 0 0 1 0 1 2 2 1 2 ...
## $ ph.karno : num 90 90 90 90 100 50 70 60 70 70 ...
## $ pat.karno: num 100 90 90 60 90 80 60 80 80 70 ...
## $ meal.cal : num 1175 1225 NA 1150 NA ...
## $ wt.loss : num NA 15 15 11 0 0 10 1 16 34 ...31 Cox回归校准曲线绘制
前面我们已经讲过逻辑回归模型的校准曲线的画法,这次我们学习cox回归的校准曲线画法。
31.1 准备数据
使用自带数据lung数据集进行演示。
这个是关于肺癌的生存数据,一共有228行,10列,其中time是生存时间,单位是天,status是生存状态,1是删失,2是死亡。其余变量是自变量,意义如下:
inst:机构代码,对于我们这次建模没啥用age:年龄sex:1是男性,2是女性ph.ecog:ECOG评分。0=无症状,1=有症状但完全可以走动,2=每天<50%的时间在床上,3=在床上>50%的时间但没有卧床,4=卧床不起ph.karno:医生评的KPS评分,范围是0-100,得分越高,健康状况越好,越能忍受治疗给身体带来的副作用。pat.karno:患者自己评的KPS评分meal.cal:用餐时消耗的卡路里wt.loss:过去6个月的体重减少量,单位是磅
大多数情况下都是使用1代表死亡,0代表删失,这个数据集用2代表死亡,但有的R包会报错,需要注意!
我们这里给它改过来:
library(dplyr)
library(tidyr)
lung <- lung %>%
mutate(status=ifelse(status == 2,1,0))
str(lung)
## 'data.frame': 228 obs. of 10 variables:
## $ inst : num 3 3 3 5 1 12 7 11 1 7 ...
## $ time : num 306 455 1010 210 883 ...
## $ status : num 1 1 0 1 1 0 1 1 1 1 ...
## $ age : num 74 68 56 57 60 74 68 71 53 61 ...
## $ sex : num 1 1 1 1 1 1 2 2 1 1 ...
## $ ph.ecog : num 1 0 0 1 0 1 2 2 1 2 ...
## $ ph.karno : num 90 90 90 90 100 50 70 60 70 70 ...
## $ pat.karno: num 100 90 90 60 90 80 60 80 80 70 ...
## $ meal.cal : num 1175 1225 NA 1150 NA ...
## $ wt.loss : num NA 15 15 11 0 0 10 1 16 34 ...然后把数据划分为训练集、测试集,下面这个划分方法是错的哈,这个示例数据样本量太少了,我想让训练集和测试集样本量都尽量多一点,所以用了一个错误的方法进行演示:
set.seed(123)
ind1 <- sample(1:nrow(lung),nrow(lung)*0.7)
train_df <- lung[ind1,]
set.seed(563435)
ind2 <- sample(1:nrow(lung),nrow(lung)*0.7)
test_df <- lung[ind2, ]
dim(train_df)
## [1] 159 10
dim(test_df)
## [1] 159 1031.2 方法1:rms
library(rms)
# 必须先打包数据
dd <- datadist(train_df)
options(datadist = "dd")
units(train_df$time) <- "day" # 单位设置为:天构建cox比例风险模型。rms可以使用内部重抽样的方法绘制校准曲线,可以选择bootstrap法或者交叉验证法,下面我们选择500次bootstrap的内部验证方法,计算时间点为第1年的校准曲线(样本太少,给警告了):
# 建立cox回归模型,时间点选择1年
coxfit1 <- cph(Surv(time, status) ~ sex + ph.ecog + ph.karno + wt.loss,
data = train_df, x = T, y = T, surv = T,
time.inc = 100 # 100天
)
# m=40表示以40个样本为1组,一般取4-6组,我们这个数据样本量太少了
# u=100和上面的time.inc对应
cal1 <- calibrate(coxfit1, cmethod = "KM", method = "boot",
u = 100, m = 40, B = 500)
## Using Cox survival estimates at 100 days31.2.1 训练集
接下来就是画图,可以直接使用plot()函数:
plot(cal1)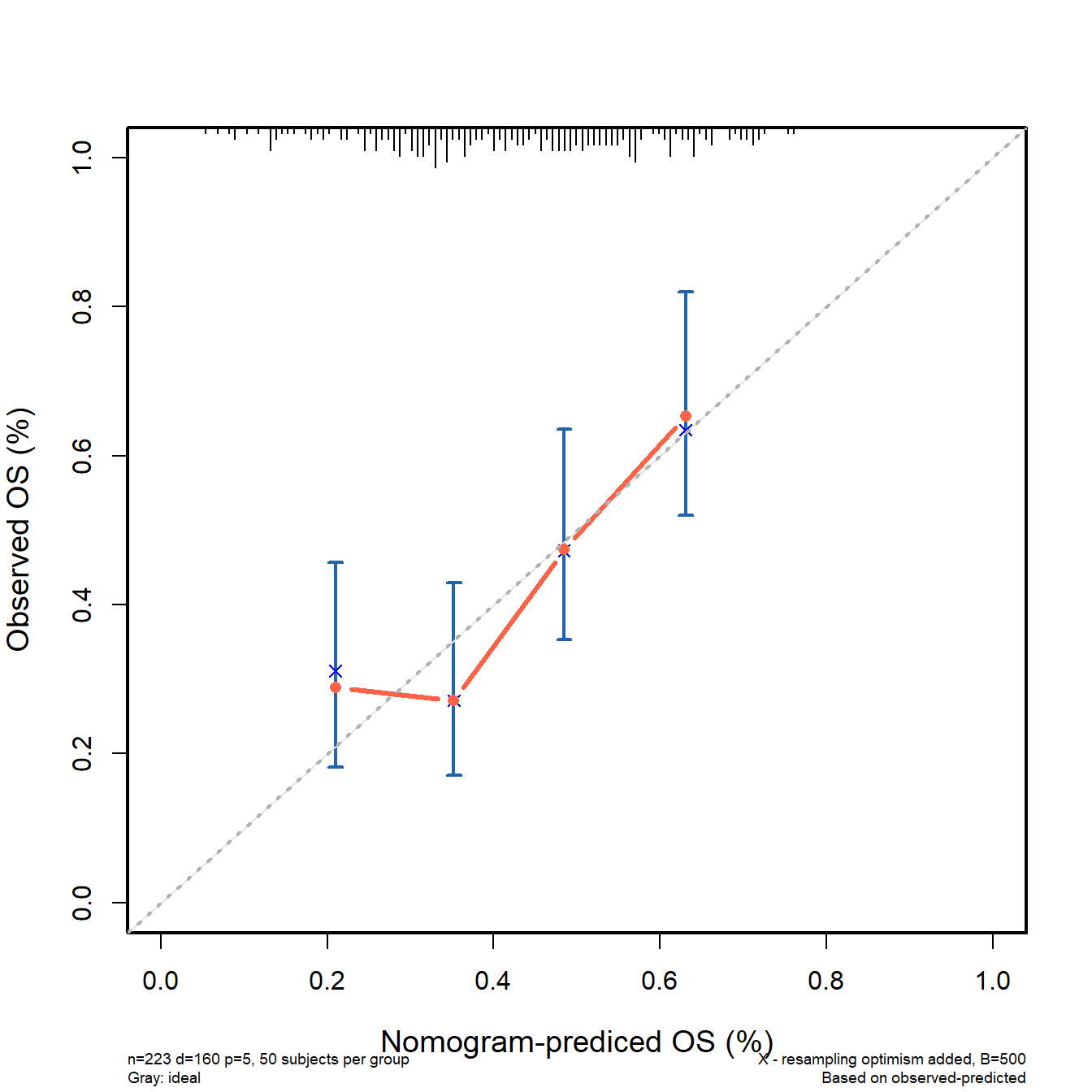
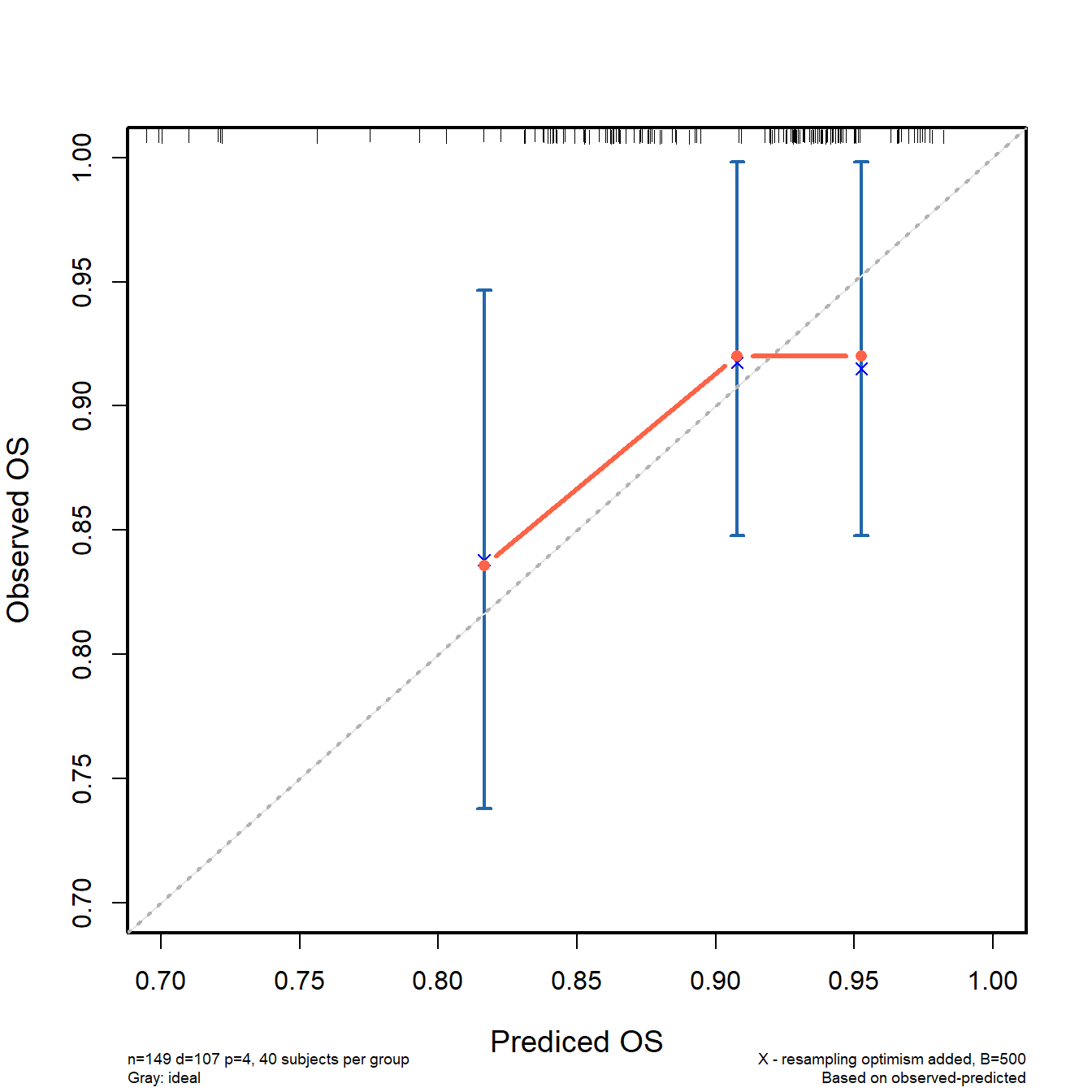
也可以提取数据,自己画,以实现更多的细节控制:
plot(cal1,
lwd = 2, # 误差线粗细
lty = 1, # 误差线类型,可选0-6
errbar.col = c("#2166AC"), # 误差线颜色
xlim = c(0.7,1),ylim= c(0.7,1), # 坐标轴范围
xlab = "Prediced OS",ylab = "Observed OS",
cex.lab=1.2, cex.axis=1, cex.main=1.2, cex.sub=0.6) # 字体大小
lines(cal1[,c('mean.predicted',"KM")],
type = 'b', # 连线的类型,可以是"p","b","o"
lwd = 3, # 连线的粗细
pch = 16, # 点的形状,可以是0-20
col = "tomato") # 连线的颜色
box(lwd = 2) # 边框粗细
abline(0,1,lty = 3, # 对角线为虚线
lwd = 2, # 对角线的粗细
col = "grey70" # 对角线的颜色
) 
31.2.2 测试集
然后是外部验证集的校准曲线:
# 获取测试集的预测的生存概率,这一步有没有都行
estimates <- survest(coxfit1,newdata=test_df,times=100)$surv
vs <- val.surv(coxfit1, newdata = test_df,
S = Surv(test_df$time,test_df$status),
est.surv = estimates,# 这一步有没有都行
u = 100 # 时间点,也是选100天
)
plot(vs)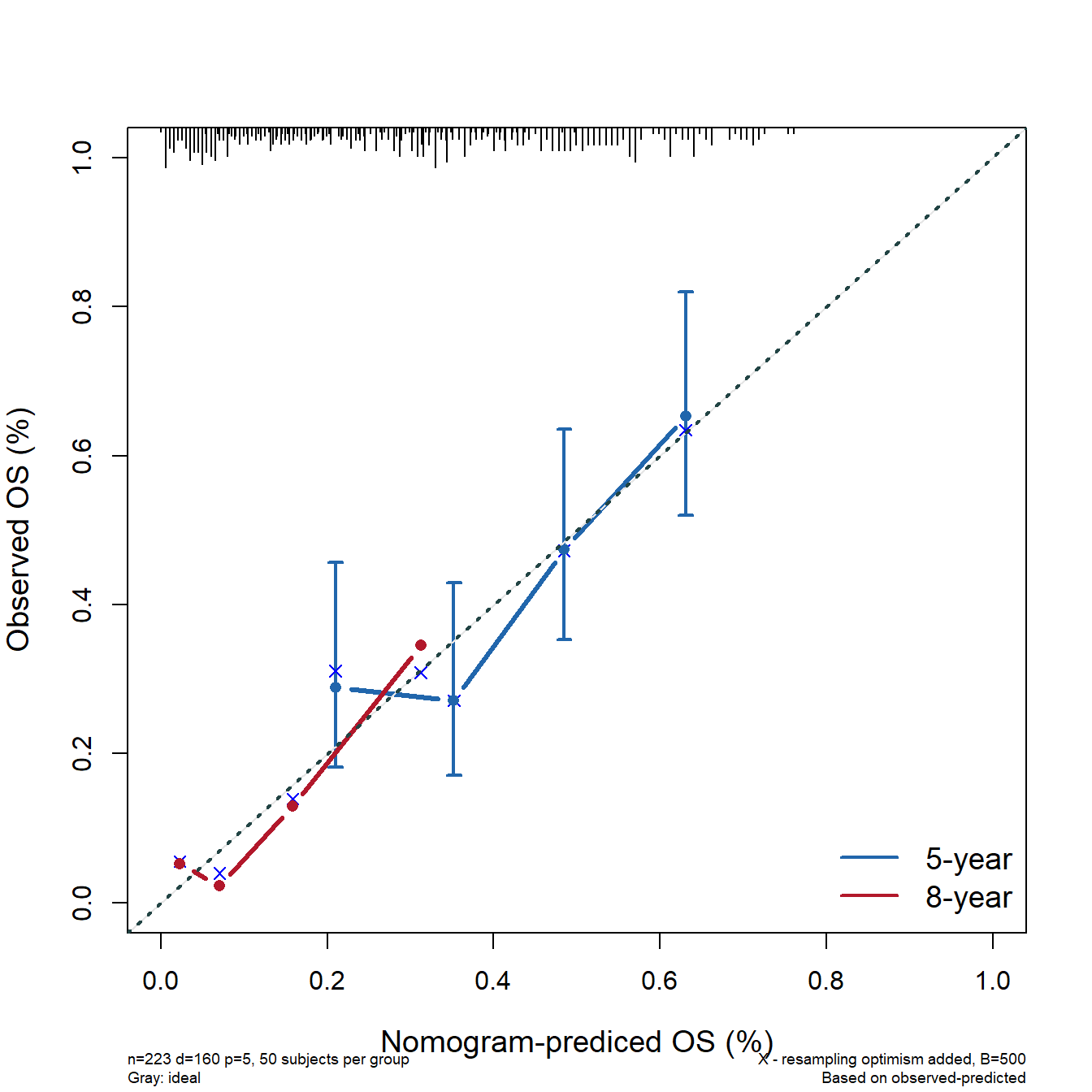
31.3 方法2:riskRegression
这个R包也非常好用,但是这种方法不能有缺失值,所以我先把缺失值去掉,然后再划分训练集和测试集,但是由于样本量太少,这里的划分方法是不正确的哈。
# 删除缺失值
df2 <- na.omit(lung)
# 划分数据
set.seed(123)
ind1 <- sample(1:nrow(df2),nrow(df2)*0.9)
train_df <- df2[ind1,]
set.seed(563435)
ind2 <- sample(1:nrow(df2),nrow(df2)*0.9)
test_df <- df2[ind2, ]
dim(train_df)
## [1] 150 10
dim(test_df)
## [1] 150 10
# 构建模型
cox_fit1 <- coxph(Surv(time, status) ~ sex + ph.ecog + ph.karno,
data = train_df,x = T, y = T)31.3.1 训练集
# 画图
library(riskRegression)
set.seed(1)
cox_fit_s <- Score(list("fit1" = cox_fit1),
formula = Surv(time, status) ~ 1,
data = train_df,
plots = "calibration",
conf.int = T,
B = 500,
M = 50, # 每组的人数
times=c(100) # 时间点选100天
)
plotCalibration(cox_fit_s,cens.method="local",# 减少输出日志
xlab = "Predicted Risk",
ylab = "Observerd RISK")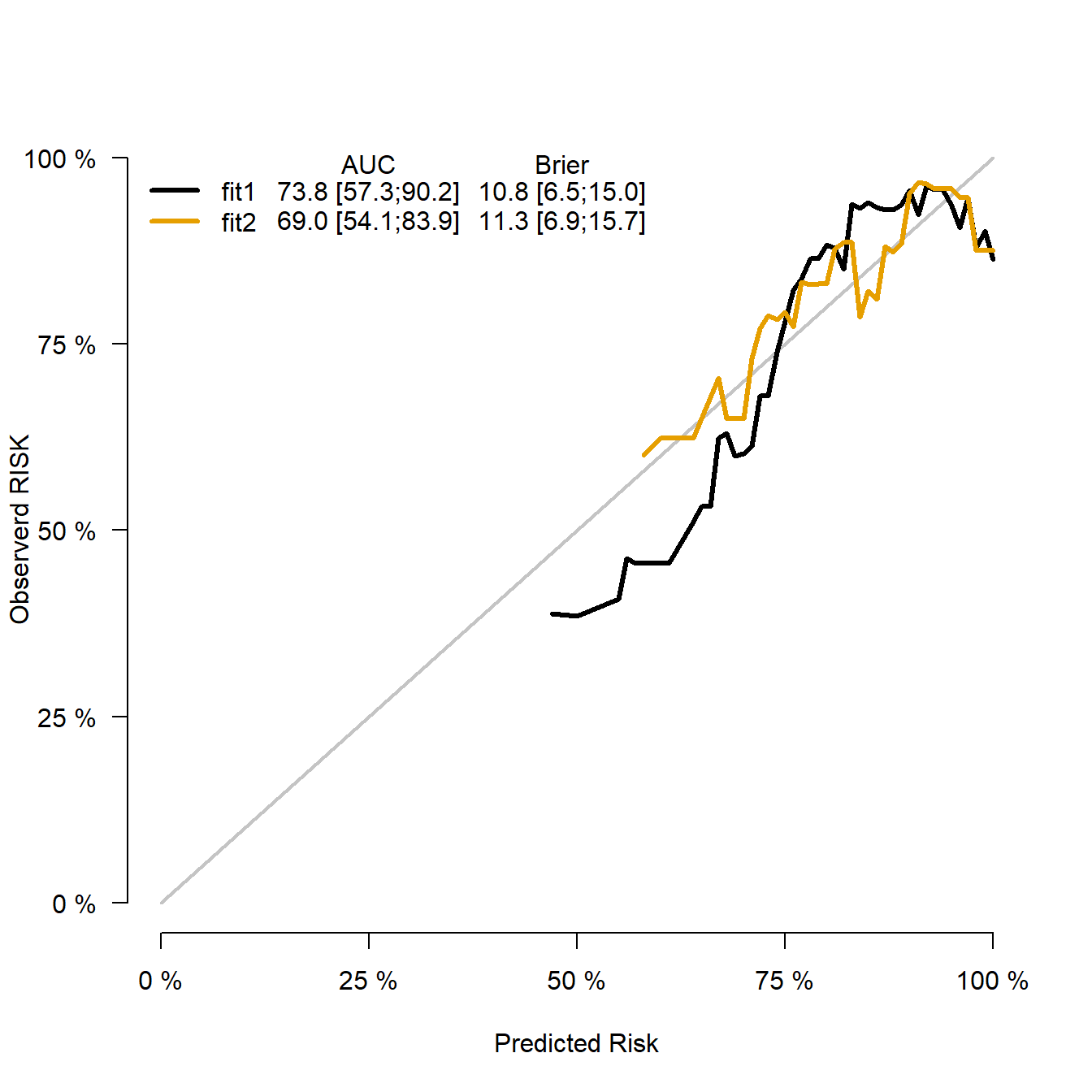
当然也是可以用ggplot2画图的。
# 获取数据
data_all <- plotCalibration(cox_fit_s,plot = F,cens.method="local")
# 数据转换
plot_df <- data_all$plotFrames$fit1
# 画图
library(ggplot2)
ggplot(plot_df, aes(Pred,Obs))+
geom_point()+
geom_line(linewidth=1.2)+
scale_x_continuous(limits = c(0,0.5),name = "Predicted Risk")+
scale_y_continuous(limits = c(0,0.5),name = "Observerd Risk")+
geom_abline(slope = 1,intercept = 0,lty=2)+
theme_bw()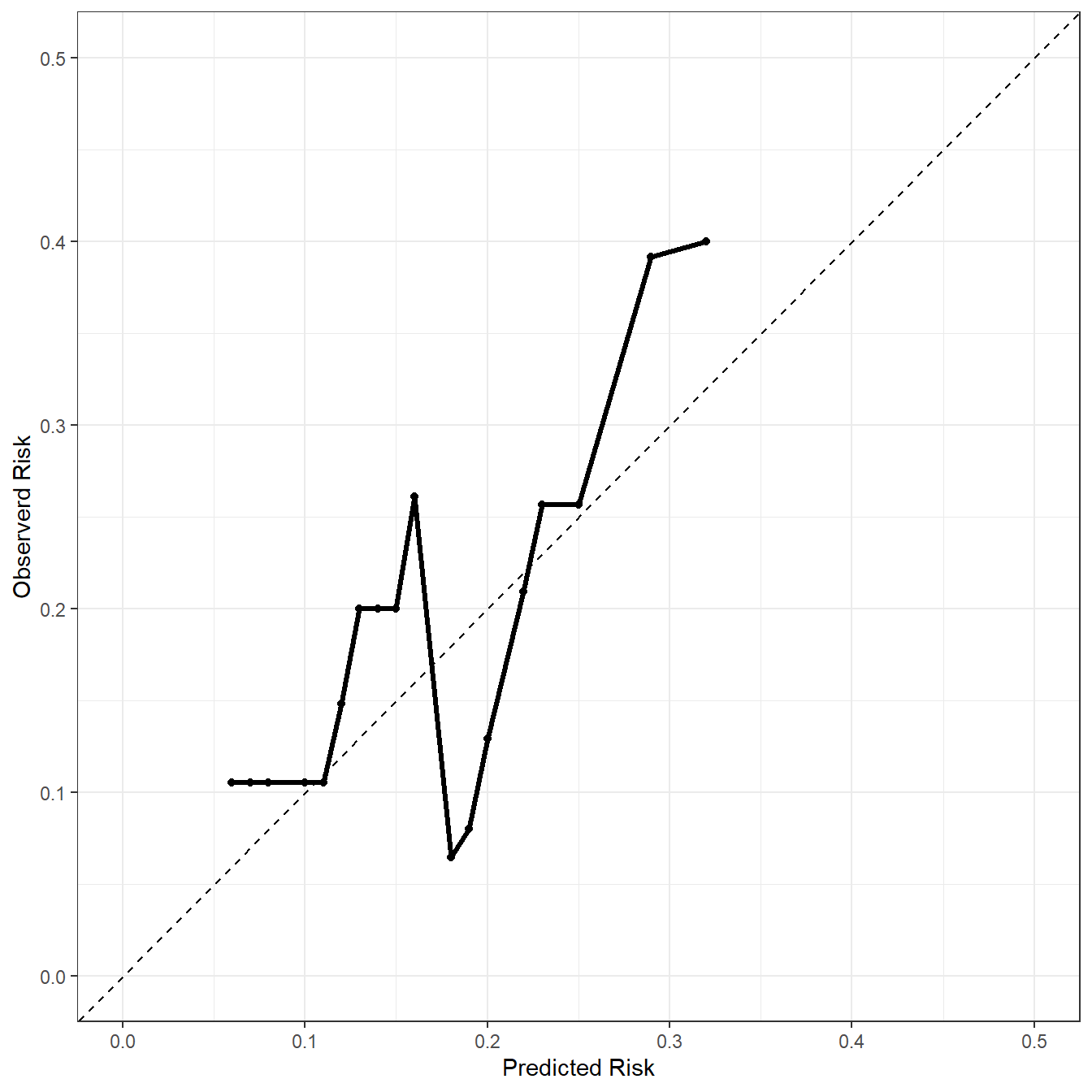
31.3.2 测试集
使用起来完全一样,只需要提供测试集即可:
set.seed(1)
cox_fit_s <- Score(list("fit1" = cox_fit1),
formula = Surv(time, status) ~ 1,
data = test_df, # 测试集
plots = "calibration",
B = 500,
M = 50,
times=c(100) # 时间点
)
plotCalibration(cox_fit_s,cens.method="local",# 减少输出日志
xlab = "Predicted Risk",
ylab = "Observerd Risk")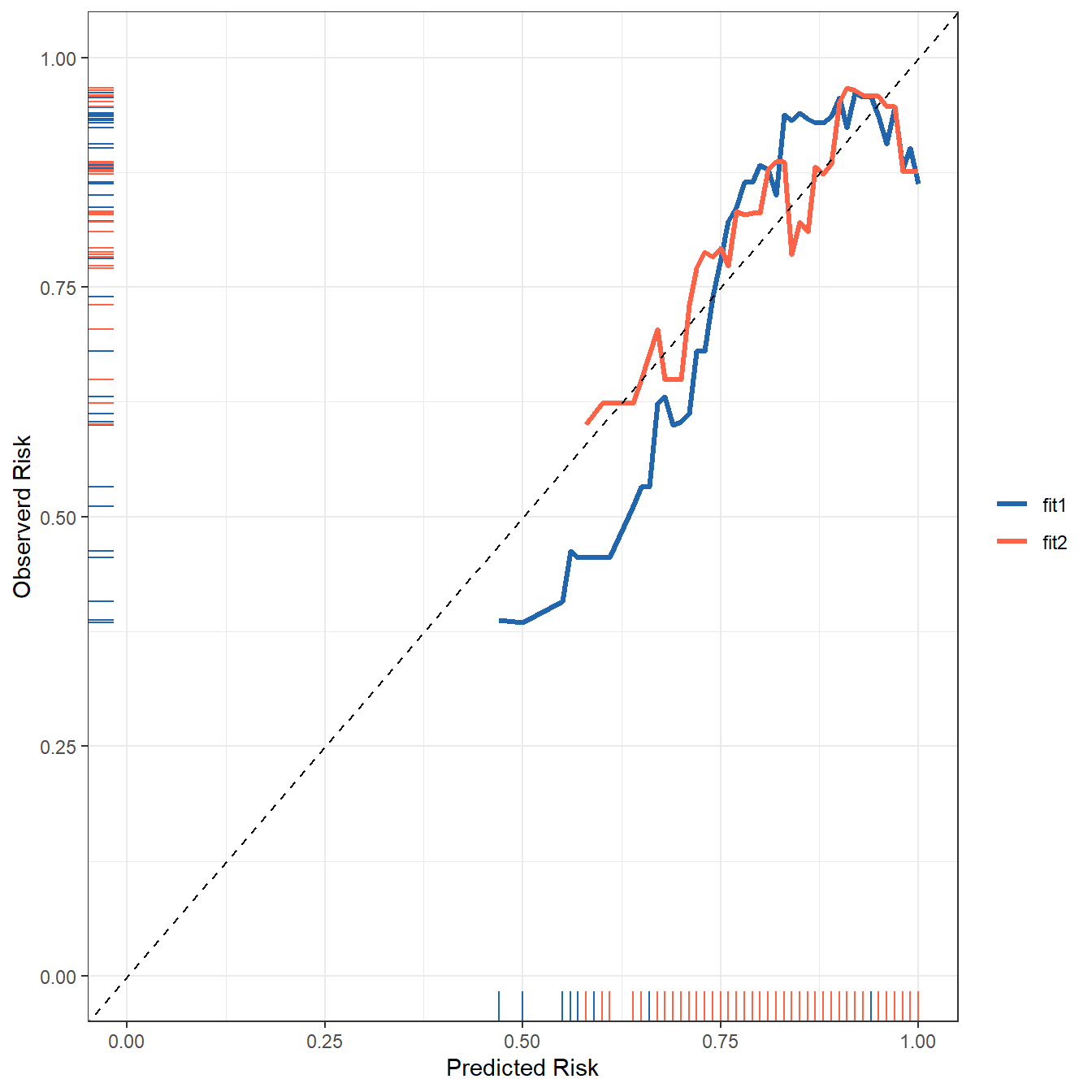
这个结果也是可以用ggplot2绘制的,就不再重复了。