rm(list = ls())
library(survival)
# 只使用部分数据
dat <- pbc[1:312,]
dat <- dat[ dat$time > 2000 | (dat$time < 2000 & dat$status == 2), ]
str(dat) # 数据长这样
## 'data.frame': 232 obs. of 20 variables:
## $ id : int 1 2 3 4 6 8 9 10 11 12 ...
## $ time : int 400 4500 1012 1925 2503 2466 2400 51 3762 304 ...
## $ status : int 2 0 2 2 2 2 2 2 2 2 ...
## $ trt : int 1 1 1 1 2 2 1 2 2 2 ...
## $ age : num 58.8 56.4 70.1 54.7 66.3 ...
## $ sex : Factor w/ 2 levels "m","f": 2 2 1 2 2 2 2 2 2 2 ...
## $ ascites : int 1 0 0 0 0 0 0 1 0 0 ...
## $ hepato : int 1 1 0 1 1 0 0 0 1 0 ...
## $ spiders : int 1 1 0 1 0 0 1 1 1 1 ...
## $ edema : num 1 0 0.5 0.5 0 0 0 1 0 0 ...
## $ bili : num 14.5 1.1 1.4 1.8 0.8 0.3 3.2 12.6 1.4 3.6 ...
## $ chol : int 261 302 176 244 248 280 562 200 259 236 ...
## $ albumin : num 2.6 4.14 3.48 2.54 3.98 4 3.08 2.74 4.16 3.52 ...
## $ copper : int 156 54 210 64 50 52 79 140 46 94 ...
## $ alk.phos: num 1718 7395 516 6122 944 ...
## $ ast : num 137.9 113.5 96.1 60.6 93 ...
## $ trig : int 172 88 55 92 63 189 88 143 79 95 ...
## $ platelet: int 190 221 151 183 NA 373 251 302 258 71 ...
## $ protime : num 12.2 10.6 12 10.3 11 11 11 11.5 12 13.6 ...
## $ stage : int 4 3 4 4 3 3 2 4 4 4 ...
dim(dat) # 232 20
## [1] 232 2028 综合判别改善指数IDI
综合判别改善指数(Integrated Discrimination Index,IDI),也适用于评价不同模型优劣的,比起NRI,IDI能够从整体角度对模型进行评价,和NRI一起使用效果更佳!相关概念的解读请参考“临床预测模型的评价”一章。本文主要介绍如何通过R语言计算IDI。
对于NRI和IDI的计算,以下是常见的R包总结:
| R包 | NRI | IDI | 二分类模型 | 生存模型 | 机器学习模型 |
|---|---|---|---|---|---|
| nricens | 支持 | 不支持 | 支持 | 支持 | 支持 |
| PredictABEL | 支持 | 支持 | 支持 | 不支持 | 支持 |
| survNRI | 支持 | 不支持 | 不支持 | 支持 | 不支持 |
| survIDINRI | 支持 | 支持 | 不支持 | 支持 | 不支持 |
28.1 二分类模型的IDI
二分类模型的NRI和IDI计算使用PredictABEL包,这个包在之前演示二分类模型的NRI时也演示过了,使用起来非常简单,可以同时给出NRI和IDI。
使用survival包中的pbc数据集用于演示,这是一份关于原发性硬化性胆管炎的数据,其实是一份用于生存分析的数据,是有时间变量的,但是这里我们用于演示分类模型,只要不使用time这一列就可以了。
然后就是准备计算IDI所需要的各个参数。
# 定义结局事件,0是存活,1是死亡
event <- ifelse(dat$time < 2000 & dat$status == 2, 1, 0)
# 建立2个模型
mstd <- glm(event ~ age + bili + albumin, family = binomial(), data = dat, x=TRUE)
mnew <- glm(event ~ age + bili + albumin + protime, family = binomial(), data = dat, x=TRUE)
# 取出模型预测概率
p.std <- mstd$fitted.values
p.new <- mnew$fitted.values在建立模型时可以选择任何能够计算概率的模型,并不一定需要是逻辑回归模型,随机森林、支持向量机等机器学习模型也都可以的。
接下来就是使用PredictABEL计算IDI:
#install.packages("PredictABEL") #安装R包
library(PredictABEL)
dat$event <- event
reclassification(data = dat,
cOutcome = 21, # 结果变量在哪一列
predrisk1 = p.std,
predrisk2 = p.new,
cutoff = c(0,0.3,0.7,1)
)
## _________________________________________
##
## Reclassification table
## _________________________________________
##
## Outcome: absent
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 121 4 0 3
## [0.3,0.7) 1 13 1 13
## [0.7,1] 0 1 3 25
##
##
## Outcome: present
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 14 0 0 0
## [0.3,0.7) 0 18 3 14
## [0.7,1] 0 1 52 2
##
##
## Combined Data
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 135 4 0 3
## [0.3,0.7) 1 31 4 14
## [0.7,1] 0 2 55 4
## _________________________________________
##
## NRI(Categorical) [95% CI]: 0.0019 [ -0.0551 - 0.0589 ] ; p-value: 0.94806
## NRI(Continuous) [95% CI]: 0.0391 [ -0.2238 - 0.3021 ] ; p-value: 0.77048
## IDI [95% CI]: 0.0044 [ -0.0037 - 0.0126 ] ; p-value: 0.28396IDI在最后一行,同时给出了95%的可信区间和P值。重分类表格中,absent是event=0的结果,present是event=1的结果。
这个包还是很全面的,如果你的模型只涉及分类,不涉及生存分析,那么在计算NRI和IDI时我推荐你使用这个包,因为使用简单,1行代码即可得到两个结果。
测试集(验证集,外部验证集)的计算也是很简单,只要计算出概率就可以了。
我们先随机建立一个测试集:
# 取前100行作为测试集,这个方法是不正规的哈
testset <- dat[1:100,]
# 计算测试集的概率
p.std_test <- predict(mstd, newdata = testset,type = "response")
p.new_test <- predict(mnew, newdata = testset,type = "response")计算IDI即可:
reclassification(data = testset,
cOutcome = 21, # 结果变量在哪一列
predrisk1 = p.std_test,
predrisk2 = p.new_test,
cutoff = c(0,0.3,0.7,1)
)
## _________________________________________
##
## Reclassification table
## _________________________________________
##
## Outcome: absent
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 49 1 0 2
## [0.3,0.7) 0 6 1 14
## [0.7,1] 0 0 3 0
##
##
## Outcome: present
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 6 0 0 0
## [0.3,0.7) 0 7 2 22
## [0.7,1] 0 1 24 4
##
##
## Combined Data
##
## Updated Model
## Initial Model [0,0.3) [0.3,0.7) [0.7,1] % reclassified
## [0,0.3) 55 1 0 2
## [0.3,0.7) 0 13 3 19
## [0.7,1] 0 1 27 4
## _________________________________________
##
## NRI(Categorical) [95% CI]: -0.0083 [ -0.1043 - 0.0876 ] ; p-value: 0.86483
## NRI(Continuous) [95% CI]: -0.15 [ -0.5466 - 0.2466 ] ; p-value: 0.45847
## IDI [95% CI]: 7e-04 [ -0.0091 - 0.0105 ] ; p-value: 0.89138easy!结果解读也是一模一样的。
28.2 生存资料的IDI
生存资料的IDI使用survIDINRI包计算。
# 安装R包
install.packages("survIDINRI")加载R包并使用,还是用上面的pbc数据集。
rm(list = ls())
library(survival)
library(survIDINRI)
# 使用部分数据
dat <- pbc[1:312,]
dat$status <- ifelse(dat$status==2, 1, 0) # 0表示活着,1表示死亡
str(dat)
## 'data.frame': 312 obs. of 20 variables:
## $ id : int 1 2 3 4 5 6 7 8 9 10 ...
## $ time : int 400 4500 1012 1925 1504 2503 1832 2466 2400 51 ...
## $ status : num 1 0 1 1 0 1 0 1 1 1 ...
## $ trt : int 1 1 1 1 2 2 2 2 1 2 ...
## $ age : num 58.8 56.4 70.1 54.7 38.1 ...
## $ sex : Factor w/ 2 levels "m","f": 2 2 1 2 2 2 2 2 2 2 ...
## $ ascites : int 1 0 0 0 0 0 0 0 0 1 ...
## $ hepato : int 1 1 0 1 1 1 1 0 0 0 ...
## $ spiders : int 1 1 0 1 1 0 0 0 1 1 ...
## $ edema : num 1 0 0.5 0.5 0 0 0 0 0 1 ...
## $ bili : num 14.5 1.1 1.4 1.8 3.4 0.8 1 0.3 3.2 12.6 ...
## $ chol : int 261 302 176 244 279 248 322 280 562 200 ...
## $ albumin : num 2.6 4.14 3.48 2.54 3.53 3.98 4.09 4 3.08 2.74 ...
## $ copper : int 156 54 210 64 143 50 52 52 79 140 ...
## $ alk.phos: num 1718 7395 516 6122 671 ...
## $ ast : num 137.9 113.5 96.1 60.6 113.2 ...
## $ trig : int 172 88 55 92 72 63 213 189 88 143 ...
## $ platelet: int 190 221 151 183 136 NA 204 373 251 302 ...
## $ protime : num 12.2 10.6 12 10.3 10.9 11 9.7 11 11 11.5 ...
## $ stage : int 4 3 4 4 3 3 3 3 2 4 ...构建参数需要的值:
# 两个只由预测变量组成的矩阵
z.std <- as.matrix(subset(dat, select = c(age, bili, albumin)))
z.new <- as.matrix(subset(dat, select = c(age, bili, albumin, protime)))然后使用IDI.INF()函数计算IDI:
res <- IDI.INF(indata = dat[,c(2,3)],
covs0 = z.std,
covs1 = z.new,
t0 = 2000, # 时间点
npert = 500, # 重抽样次数
seed1 = 1234 # 设定重抽样的种子数
)
IDI.INF.OUT(res) # 提取结果
## Est. Lower Upper p-value
## M1 0.020 -0.004 0.055 0.104
## M2 0.202 -0.064 0.382 0.084
## M3 0.011 -0.003 0.033 0.088- m1:IDI的值,可信区间,P值
- m2:NRI的值,可信区间,P值
- m3:风险分数的中位数提升(median improvement of risk score),可信区间,p值。
这里的NRI是连续型NRI,定义是当“1/2 NRI(>0)”时的值,详细内容请参考该函数的帮助文档。
计算IDI也是基于概率的,这个函数默认会帮你建立cox模型从而计算生存概率,所以这个函数计算的其实是两个cox模型的IDI。
除此之外这个包还可以绘制图形,以图形的方式同时展示IDI、NRI、中位数提升分数:
IDI.INF.GRAPH(res)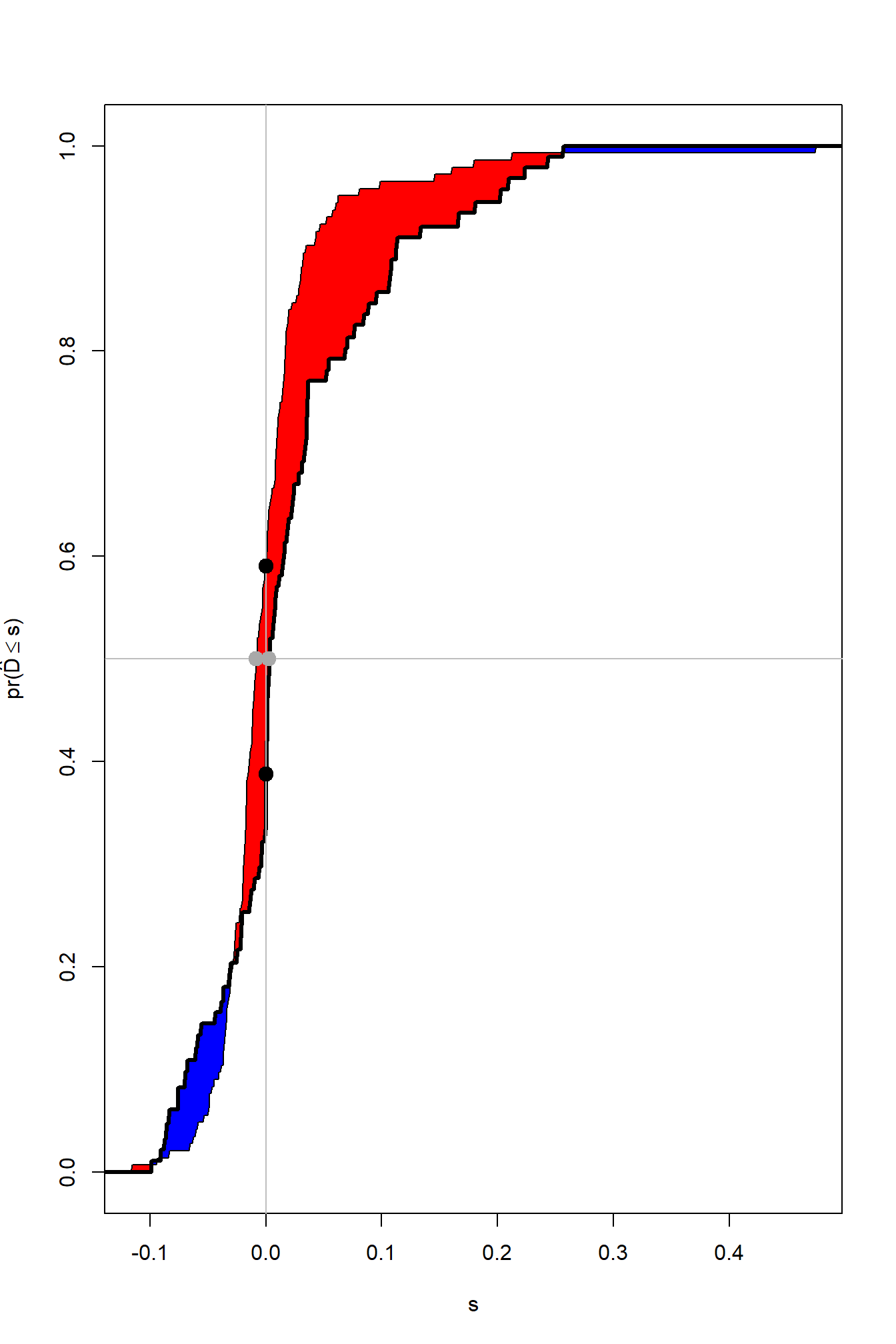
红色面积减去蓝色面积得到的值就是IDI;两个黑点之间的垂直距离就是连续NRI;两个灰点之间的距离是中位数提升。
由于这个函数计算IDI时不支持使用概率,只能使用原始数据,所以理论上这个函数无法计算测试集(验证集,外部验证集)的IDI,而且也不支持计算机器学习模型(比如随机生存森林、生存支持向量机等)的IDI。目前我并没有发现其他更好的R包,大家有知道的欢迎评论区留言。
但是在实际使用时,NRI和IDI都是作为比较最终模型准确性的,所以通常都是只做一遍就可以了,不需要再训练集、验证集各来一遍。