library(survival)
library(rms)
rm(list = ls())
dim(lung)
## [1] 228 10
str(lung)
## 'data.frame': 228 obs. of 10 variables:
## $ inst : num 3 3 3 5 1 12 7 11 1 7 ...
## $ time : num 306 455 1010 210 883 ...
## $ status : num 2 2 1 2 2 1 2 2 2 2 ...
## $ age : num 74 68 56 57 60 74 68 71 53 61 ...
## $ sex : num 1 1 1 1 1 1 2 2 1 1 ...
## $ ph.ecog : num 1 0 0 1 0 1 2 2 1 2 ...
## $ ph.karno : num 90 90 90 90 100 50 70 60 70 70 ...
## $ pat.karno: num 100 90 90 60 90 80 60 80 80 70 ...
## $ meal.cal : num 1175 1225 NA 1150 NA ...
## $ wt.loss : num NA 15 15 11 0 0 10 1 16 34 ...14 计算列线图得分及危险分层
列线图是模型的图形化表达方式,通过列线图可以计算直观的看出某个患者的得分,方便分析患者的风险。关于列线图,我们介绍过很多次了,公众号后台回复列线图即可获取合集链接。
前面一篇介绍了列线图增加彩色风险分层条带
那么,如何根据列线图得分进行危险分层?其实思路很简单,先计算每个患者的得分,然后根据最佳截断值进行分层即可。这个最佳截断值,我们也介绍过非常多的方法了,公众号后台回复最佳截点,即可获取合集链接:
14.1 准备数据
使用R包自带数据。
14.2 建立模型和列线图
使用rms包构建模型和列线图。
大多数情况下都是使用1代表死亡,0代表删失,这个数据集用2代表死亡。在这里没有影响,但有的R包会报错,需要注意!
dd <- datadist(lung)
options(datadist = "dd")构建cox比例风险模型:
coxfit <- cph(Surv(time, status) ~ age + sex + ph.ecog + ph.karno + pat.karno,
data = lung ,surv = T)
# 构建生存函数,注意你的最大生存时间
surv <- Survival(coxfit)
surv1 <- function(x) surv(365,x) # 1年OS
surv2 <- function(x) surv(365*2,x) # 2年OS
nom <- nomogram(coxfit,
fun = list(surv1,surv2),
lp = T,
funlabel = c('1-year survival Probability',
'2-year survival Probability'),
maxscale = 100,
fun.at = c(0.95,0.9,0.8,0.7,0.6,0.5,0.4,0.3,0.2,0.1))然后就是画图:
plot(nom,
lplabel="Linear Predictor",
xfrac = 0.2, # 左侧标签距离坐标轴的距离
#varname.label = TRUE,
tcl = -0.2, # 刻度长短和方向
lmgp = 0.1, # 坐标轴标签距离坐标轴远近
points.label ='Points',
total.points.label = 'Total Points',
cap.labels = FALSE,
cex.var = 1, # 左侧标签字体大小
cex.axis = 1, # 坐标轴字体大小
col.grid = gray(c(0.8, 0.95))) # 竖线颜色
到这里都很简单。
14.3 计算分数
使用nomogramFormula计算每个患者的列线图得分。
两种方法,一种是使用formula_lp根据线性预测值计算,另一种是使用formula_rd根据原始数据(raw_data)计算,两种方法结果差不多,任选一种即可。
library(nomogramFormula)
results <- formula_lp(nomogram = nom)
points1 <- points_cal(formula = results$formula, lp = coxfit$linear.predictors)
#或者
#results <- formula_rd(nomogram = nom)
#points1 <- points_cal(formula = results$formula, rd = lung)
length(points1)
## [1] 223
head(points1)
## 1 2 3 4 5 6
## 129.96853 98.56938 90.51815 142.40181 102.54570 104.51291这样每个人都根据列线图得到一个分数,根据这个分数就可以把所有患者分成高风险组/低风险组了。
14.4 分层
假如我们想根据列线图得分进行分层,使得分层后两组的K-M生存分析的p值最小,方法很多,任选一种即可,我这里就用surv_cutpoint演示。
但是计算出来的分数有223个,原始数据是228个,因为数据有缺失值,在建立模型时有5个样本被删了,这时候你回过去找不一定找得到缺失值在哪(我能找到),所以建议一开始就把缺失值处理掉。
library(tidyr)
library(survminer)
# 去掉缺失值
tmp <- lung %>%
drop_na(ph.ecog,ph.karno,pat.karno)
dim(tmp)
## [1] 223 10
tmp$points <- points1
# 确定最佳截点,然后根据最佳截点分层
res.cut <- surv_cutpoint(tmp, time = "time", event = "status",
variables = "points"
)
# 查看最佳分割点
res.cut[["cutpoint"]][["cutpoint"]]
## [1] 109.2188
# 根据最佳截点分层
res.cat <- surv_categorize(res.cut)绘制生存曲线:
library("survival")
fit <- survfit(Surv(time, status) ~ points, data = res.cat)
ggsurvplot(fit, data = res.cat, pval = T)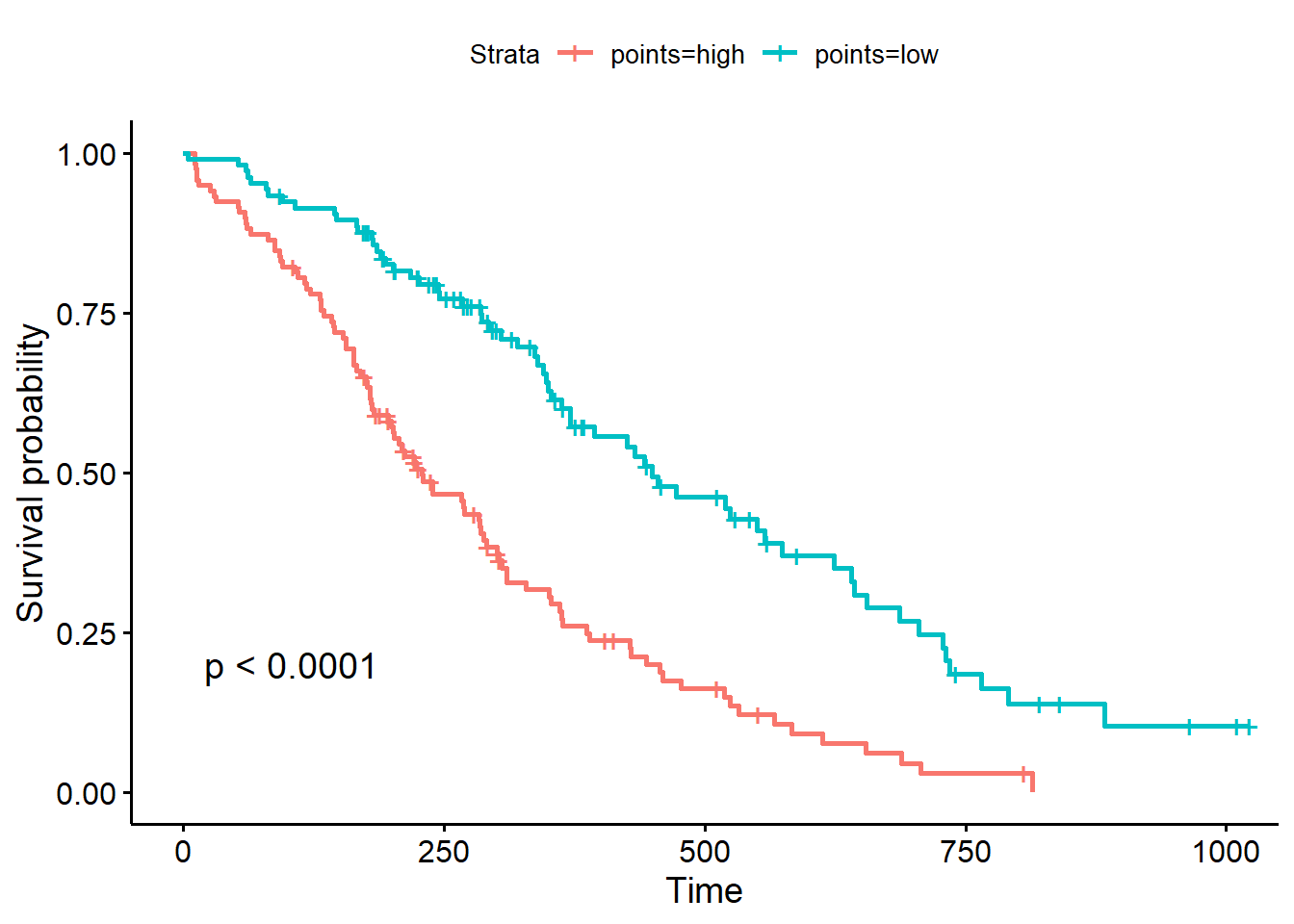
中间的数据展示省略了很多,还不熟悉这一套流程的可以一步一步的看,结合之前的文章。
14.5 外部验证集
大家一定要学会变通啊,学会R语言的基础知识,才好举一反三。
外部验证集的分数计算直接使用formula_rd()函数就可以了(不管训练集还是外部验证集我都推荐使用这个,前面为了演示没用这个函数),rd参数提供外部验证集即可:
# 假设这是外部验证集
valdf <- na.omit(lung[1:100,])
# 计算分数
results <- formula_rd(nomogram = nom)
points_val <- points_cal(formula = results$formula,
rd = valdf) # 外部验证集
length(points_val)
## [1] 74
head(points_val)
## [1] 98.56934 142.40178 104.51289 131.33431 112.08372 109.11084前面我们计算的最佳截点是109.2188,现在就根据这个截点对外部验证集分组,高于这个值就是高风险,低于这个值就是低风险:
valdf$points <- points_val
valdf$groups <- ifelse(valdf$points>109.2188,"high","low")
head(valdf[,11:12])
## points groups
## 2 98.56934 low
## 4 142.40178 high
## 6 104.51289 low
## 7 131.33431 high
## 8 112.08372 high
## 9 109.11084 low绘制外部验证集的生存曲线:
library("survival")
fit <- survfit(Surv(time, status) ~ groups, data = valdf)
ggsurvplot(fit, data = valdf, pval = T)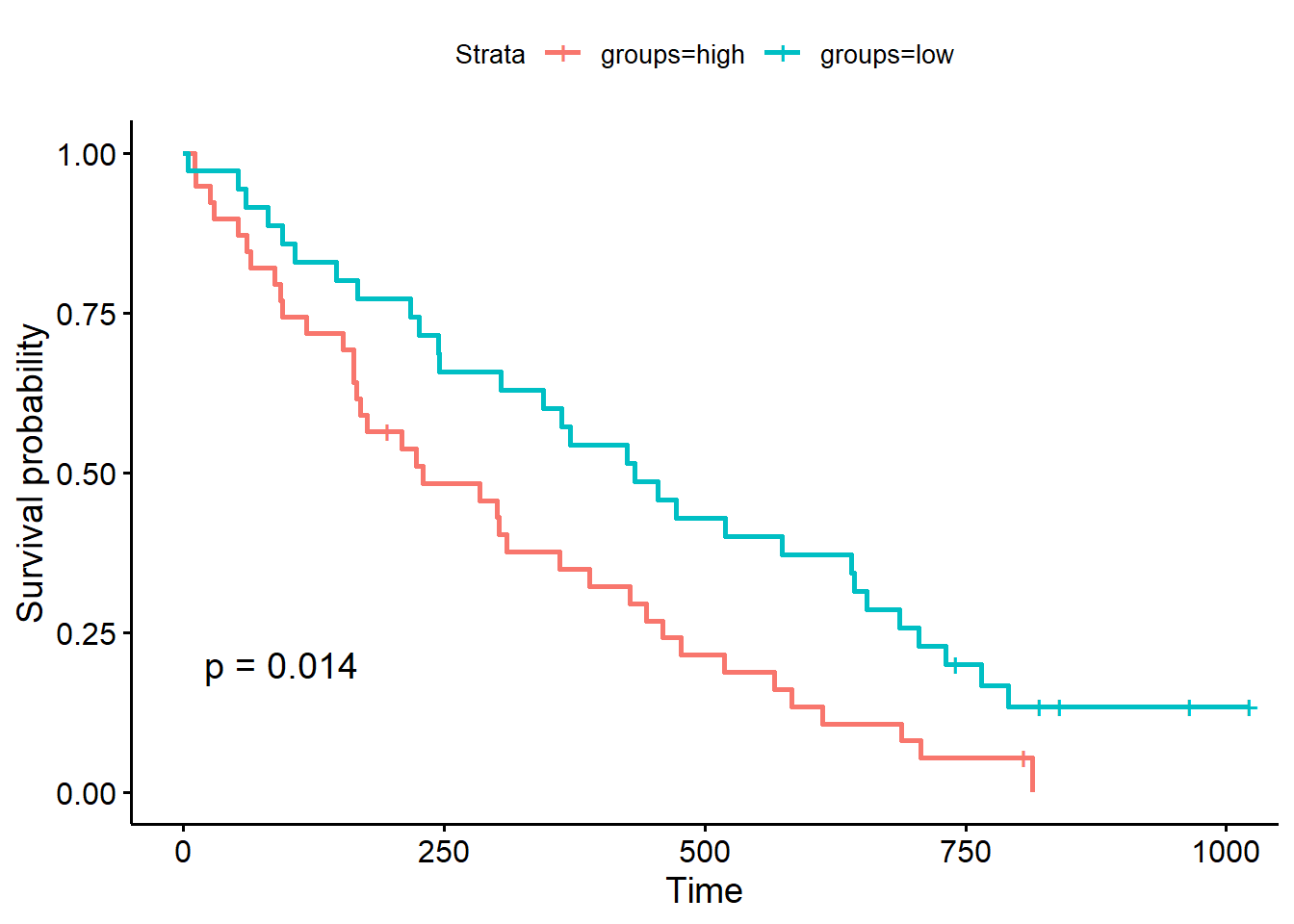
是不是一样的简单?
14.6 扩展
这里是根据列线图的得分进行分层的,其实也可以直接根据模型得到的线性预测值进行分层,就是直接使用predict即可:
predict(coxfit,head(lung))
## 1 2 3 4 5 6
## 0.3113300 -0.2213878 -0.3579849 0.5222729 -0.1539256 -0.1205499这个东西就是大家常见的risk-score,当然这只是其中一种计算方式,不同的模型计算方法略有不同。
而且cox回归得到的这个线性预测值又叫做预后指数(prognosis index, PI),这个值在统计学中是有明确含义的,根据这个值进行危险分层也是完全没问题的。
预后指数越大,患者风险越大,预后越差。–孙振球《医学统计学》第4版P293
最早的建模类文章都是这么干的,现在也不少见。优点就是少了计算分数那一步,缺点嘛暂时没发现,毕竟都是模仿,你发文章只要把你的故事说清楚即可。